| Autore |
Discussione |
|
|
Lindbergh
Nuovo Arrivato

7 Messaggi |
 Inserito il - 24 ottobre 2009 : 13:32:45 Inserito il - 24 ottobre 2009 : 13:32:45




|
Cerco risposta alla seguente domanda in cambio di un semplice GRAZIE!
Come si determinano gli intervalli nucleotidici ("nucleotide spans") tra le sequenze note di DNA ribosomiale fungino comprendente in successione le sequenze: 1) gene RNA 18S (parziale), 2) ITS1, 3) gene RNA 5.8S, 4) sequenza ITS2 e 5) gene RNA 28S (parziale)?
In altri termini come č possibile identificare le sequenze esatte delle regioni ITS1 e ITS2 che sono frapposte alle sequenze 18S (parziale), 5.8S (completa) e 28S (parziale)?
Saluti
|
|
|
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Cittā: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 25 ottobre 2009 : 14:46:31 Inserito il - 25 ottobre 2009 : 14:46:31





|
| mmm non č molto chiara la domanda, ma immagino che sia innanzitutto necessario avere la sequenza completa del DNA ribosomiale del fungo/ceppo che vuoi studiare, dopodichč puoi controllare tra le annotazioni dello stesso, o fare un allineamento sulle sequenze del 18S e 5.8S per trovare la sequenza ITS1 che si trova in mezzo. Perdona la mia ignoranzia, ma queste sequenze sono semplicemente composte da tutto ciķ che si trova tra il 18S e 5.8S (e 28S), oppure sono sottosequenze di queste? |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
mak_steon
Utente Junior


138 Messaggi |
 Inserito il - 26 ottobre 2009 : 09:12:19 Inserito il - 26 ottobre 2009 : 09:12:19




|
Potresti amplificare la zona di interesse utilizzando primer esterni e poi sequenziare l'amplicone.
Ciao ciao |
 |
|
|
Lindbergh
Nuovo Arrivato

7 Messaggi |
 Inserito il - 26 ottobre 2009 : 16:49:28 Inserito il - 26 ottobre 2009 : 16:49:28




|
Citazione:
Messaggio inserito da dallolio_gm
mmm non č molto chiara la domanda, ma immagino che sia innanzitutto necessario avere la sequenza completa del DNA ribosomiale del fungo/ceppo che vuoi studiare, dopodichč puoi controllare tra le annotazioni dello stesso, o fare un allineamento sulle sequenze del 18S e 5.8S per trovare la sequenza ITS1 che si trova in mezzo. Perdona la mia ignoranzia, ma queste sequenze sono semplicemente composte da tutto ciķ che si trova tra il 18S e 5.8S (e 28S), oppure sono sottosequenze di queste?
Scusami se non sono stato molto chiaro ma come ho scritto sopra la sequenza č nota. Purtroppo per questa specie fungina non ci sono sequenze simili depositate in GenBank. E' possibile identificare le sequenze delle regioni ITS1 e ITS2 del rDNA? Come si puō vedere dall'immagine allegata la sequenza ITS1 si trova tra le sequenze 18S e 5.8S, mentre la sequenza ITS2 si trova tra la 5.8S e la 28S.
Grazie
Immagine:
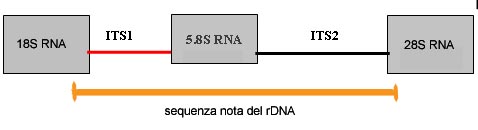
19,91 KB |
 |
|
|
Lindbergh
Nuovo Arrivato

7 Messaggi |
 Inserito il - 26 ottobre 2009 : 17:48:16 Inserito il - 26 ottobre 2009 : 17:48:16




|
Citazione:
Messaggio inserito da mak_steon
Potresti amplificare la zona di interesse utilizzando primer esterni e poi sequenziare l'amplicone.
Ciao ciao
Giā fatto! Come faccio a distinguere le sequenze ITS1 e ITS2 dall'amplicone sequenziato?
Ciao |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Cittā: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 27 ottobre 2009 : 14:21:57 Inserito il - 27 ottobre 2009 : 14:21:57





|
Prova a controllare su fungalgenomes.org:
- http://fungalgenomes.org/
- http://fungalgenomes.org/gb/gbrowse/Browser_tracks/
Io proverei a prendere le sequenze del 18S, 5.8S e 28S e allinearle sulla tua sequenza... le parti che non si allineano dovrebbero corrispondere a ITS1 e IST2. |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
Lindbergh
Nuovo Arrivato

7 Messaggi |
 Inserito il - 28 ottobre 2009 : 19:18:31 Inserito il - 28 ottobre 2009 : 19:18:31




|
Citazione:
Messaggio inserito da dallolio_gm
Prova a controllare su fungalgenomes.org:
- http://fungalgenomes.org/
- http://fungalgenomes.org/gb/gbrowse/Browser_tracks/
Io proverei a prendere le sequenze del 18S, 5.8S e 28S e allinearle sulla tua sequenza... le parti che non si allineano dovrebbero corrispondere a ITS1 e IST2.
Innanzitutto grazie per l'interesse.
La soluzione che proponi č elegante, tuttavia non risolvo il problema perchč il genoma della specie di cui conosco la sequenza ITS-rDNA non č presente nel sito che hai indicato. Inoltre, per curiositā ho provato a cercare nel sito le sequenze 18S, 5.8S e 28S di un'altra specie fungina ma non sono riuscito a trovarle.
Ciao e grazie ancora |
 |
|
| |
Discussione |
|






