Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
JimmyCorgan
Nuovo Arrivato

5 Messaggi |
 Inserito il - 28 luglio 2012 : 16:41:23 Inserito il - 28 luglio 2012 : 16:41:23




|
Sto preparando un esame di biologia molecolare, ma ad un settimana dall'orale non mi sono ancora tolto un sacco di dubbi
Per esempio:
- Ricombinazione del DNA
Ricombinazione Omologa: ma quando ho risolto le giunzioni di Hollyday mi ritrovo due molecole di DNA con filamenti "mescolati". Ma vista la non-complementarietà totale delle basi (differenti alleli) non dovrebbero entrare in gioco meccanismi di riparazione del DNA? Tipo MMR o sistemi simili?
Ricombinazione sito-specifica: qualcuno che ha la pazienza di spiegarmela, e soprattutto di indicarmi le differenze che intercorrono fra questa tipologia di ricombinazione e quella dei trasposomi?
- Replicazione DNA
Ma alla fin fine (prendiamo il caso "semplice" dei procarioti). Hanno 1 origine di replicazione e due forcelle di replicazione. Per ogni forcella di replicazione quante DNA polimerasi abbiamo all'opera? (consideriamo solo DNA pol di vera e propria sintesi, non polimerasi di correzione). In certi testi dicono 1, costituita da 2 core (considerano la DNA pol III dell'E.coli), 2 subunità tao, 2 subunità gammaed altre 3/4 subunità legate alle unità beta; altri semplicemente dicono che sono 2, una per il filamento discontinuo ed una per il filamento guida.
|
|
|
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 30 luglio 2012 : 17:30:11 Inserito il - 30 luglio 2012 : 17:30:11





|
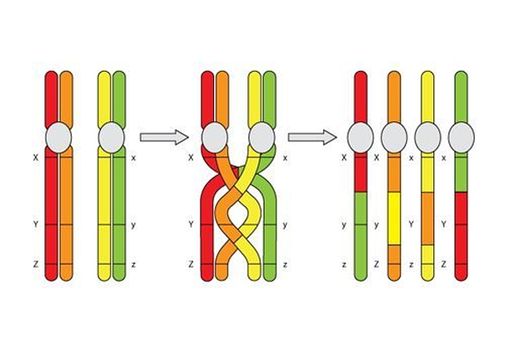
Per quanto riguarda il tuo dubbio sulla ricombinazione, la risposta è no. Hai ricombinazione in 2 casi differenti, o durante il crossing over, oppure in seguito a danno a doppio filamento al DNA (DSBs).
Durante il crossing over la ricombinazione è totale, ovvero un intero frammento del cromosoma A si ricombina col cromosoma B.
Immagine:
22,63 KB.
Quando si verifica un DSB, si generano delle estremità 3' single strand. Queste si appaiano sul filamento complementare dell'altro cromosoma, queste fanno da innesco per la sintesi del frammento perso in seguito a DSB. Per cui se l'altro cromosoma presentava un allele differente nella sequenza, ora verrà copiato dalla DNA polimerasi, per cui quando si risolveranno le giunzioni, ogni frammento si appaierà perfettamente col suo complementare. Anzi quello che può succedere in questi casi è una perdita di "eterozzigosità" nella regione ricombinata.
La ricombinazione sito specifica non è altro che una ricombinazione, la cui riuscita richiede la presenza di specifiche sequenze di DNA, solo queste e non altre sono riconosciute dall'enzima,questo infatti è in grado di catalizzare il taglio solo in queste sequenze e quindi favorire poi il fenomeno di ricombinazione, oppure una ricombinazione sito specifica è quella che avviene per ricombinazione omologa, in questo caso le sequenze di appaiamento devono essere identiche.
Oltre alla ricombinazione sito-specifica esiste la ricombinazione random, cosa che fanno i trasposoni, ovvero la ricombinazione è del tutto casuale.
Non sono entrato troppo nello specifico, Spero di essere stato chiaro, se hai dubbi chiedi pure. |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
JimmyCorgan
Nuovo Arrivato

5 Messaggi |
 Inserito il - 30 luglio 2012 : 19:03:44 Inserito il - 30 luglio 2012 : 19:03:44




|
Grazie per la risposta :)
Tuttavia non mi è ancora chiaro il meccanismo della ricombinazione omologa (premetto che non ho mai studiato genetica, quindi magari il mio è esclusivamente un problema di conoscenza preliminare).
Da quello che ho capito questa può avvenire quando due coppie di cromosomi sono omologhi, giusto? E se sono omologhi questi possono differenziare l'uno dall'altro esclusivamente per quanto riguarda l'ambito allelico, giusto ancora? Cioè nella figura che hai riportato, la sequenza rossa e quella verde sono uguali? Hai una figura che spieghi un pò più nel dettaglio il meccanismo intimo?
Grazie ancora  |
 |
|
|
roberta.s
Utente Junior


Città: Parigi

564 Messaggi |
 Inserito il - 31 luglio 2012 : 11:59:54 Inserito il - 31 luglio 2012 : 11:59:54




|
Normale che tu abbia cosi' tanti dubbi se non hai studiato genetica! su pubmed ci sono moltissime review sulla ricombinazione omologa, potresti darci un'occhiata.
In linea generale, comunque, la ricombinazione omologa avviene per riparare delle rotture a doppio filamento (ad esempio durante la meiosi, o per portare a ) e porta due cromosomi omologhi (uno materno e uno paterno) a scambiarsi "pezzi". ovviamente questi "pezzi" somo abbastanza simili da trovare omologia, ma differiscono tuttavia in alcuni punti dando origine a SNP (polimorfismi di singoli nucleotidi). (sai che il nostro genoma differeisce da un individuo all'altro).
non so quanto in là posso andare con la spiegazione, ma se ti interessa possiamo continuare a discutere. |
 |
|
|
JimmyCorgan
Nuovo Arrivato

5 Messaggi |
 Inserito il - 31 luglio 2012 : 12:36:48 Inserito il - 31 luglio 2012 : 12:36:48




|
Volentieri 
In effetti i miei dubbi (anche senza entrare nei dettagli del meccanismo intimo) erano dovuto al fatto che ragionavo in termini di scambio tra doppi filamenti, e non tra cromosomi. Infatti ragionando con questi ultimi il discorso fila. Quindi un'altra mia domanda è: la ricombinazione omologa come avviene quando si tratta di due doppi filamenti (lasciamo da parte l'ambito del DSB)? In quel caso ci deve essere un pre-appaiamento delle basi identiche tra un filamento del DNA A (per esempio) e uno dei due filamenti del DNA B con conseguente neo-sintesi dei filamenti ricombinati, giusto?
|
 |
|
|
roberta.s
Utente Junior


Città: Parigi

564 Messaggi |
 Inserito il - 31 luglio 2012 : 13:47:53 Inserito il - 31 luglio 2012 : 13:47:53




|
Guarda la figura che ti ha allegato zerhos. Si vede che ognuno dei due cromosomi omologhi è formato da due molecole di DNA. Difatti la ricombinazione omologa avviene dopo la fase S, quindi dopo che ogni molecola di DNA che costituisce ciascun cromosoma si è replicata. In sintesi, cio' che accade è:
- rottura a DOPPIO filamento del DNA di un cromosoma omologo (DSB), diciamo quello materno (nella figura di zerhos, la rottura riguarderebbe sia il filamento rosso che quello arancione del primo cromosoma che è disegnato)
- resezione delle estremità del DNA a livello del sito di danno, con conseguente formazione di un "buco", che deve essere riparato
- invasione di uno dei due filamenti del cromosoma che ha subito il danno (in questo esempio il cromosoma materno) per cercare omologia nel cromosoma omologo (ovvero nel cromosoma paterno)
- una volta trovata l'omologia, si procede con la sintesi del DNA per colmare il "buco" di cui sopra: cosi' i due filamenti materni vengono riparati usando come stampo i filamenti paterni a livello della regione di omologia
- finita la sintesi del DNA, la struttura formata da i due cromosomi omologhi (detta struttura di Holliday) puo' risolversi in due modi, per dare o meno origine a crossing over |
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 31 luglio 2012 : 18:20:14 Inserito il - 31 luglio 2012 : 18:20:14





|
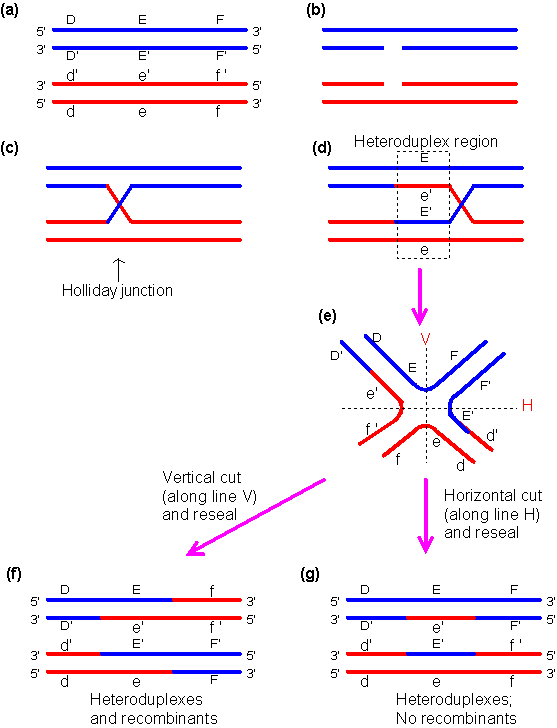
Secondo me il tuo dubbio deriva dal fatto che non hai ben capito come si risolve la giunzione di holliday nel caso di un crossing over
Andiamo per ordine:
Immagine:
10,01 KB
Come puoi vedere da questa istantanea, 2 dei 4 filamenti invadono la sequenza complementare sull'altro cromosoma, gli altri 2 sono intonsi.
A questo punto l'enzima di taglio risolve il complesso di holiday in 2 possibili modi, guardando la figura, esiste il modo f), quello a sinistra e il modo g), quello a destra.
modello g):
il taglio interessa solamente il filamento che ha iniziato l'invasione sull'altro cromosoma, in questa condizione si ripristinerà tutta la sequenza del cromosoma originario(guarda le regioni D' ed F' ad esempio) tranne che per una piccola regione centrale in genere di qualche centinaio di basi(in realtà la lunghezza deriva dalla durata dell'evento di invasione del filamento), quella regione deriva dall'altro cromosoma, per cui solo in questa regione si è in una condizione di eteroduplex. Essendo un eteroduplex, può capitare che l'omologia non sia assoluta e quindi neanche l'appaiamento dei filamenti, intervengono allora i sistemi di riparo a sistemare questa regione. Il questo caso però non si ha ricombinazione perchè la sequenza rimane invariata tranne che per quella piccola regione centrale.
Modello f):
Il taglio cade sul filamento intonso, questo fa si che l'estremità destra e l'estremità sinistra (considerato come punto centrale la regione di invasione) saranno ricombinate, nel senso che la prima parte del filamento continuerà con una seconda parte derivata dal suo omologo dell'altro cromosoma. Quì come puoi vedere quello che si sposta è l'intero duplex di DNA che da un cromosoma va all'altro, per cui l'appaiamento è perfetto e non richiede nessun sistema di riparo (tranne sempre quella piccola regine centrale eteroduplex). |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
roberta.s
Utente Junior


Città: Parigi

564 Messaggi |
 Inserito il - 31 luglio 2012 : 18:37:01 Inserito il - 31 luglio 2012 : 18:37:01




|
| Aggiungerei che, se si parla di ricombinazione omologa, c'è da precisare che essa è indotta molto più frequentemente da DSB che da single strand breaks, i quali sono solitamente riparati tramite NHEJ |
 |
|
|
roberta.s
Utente Junior


Città: Parigi

564 Messaggi |
 Inserito il - 31 luglio 2012 : 18:39:43 Inserito il - 31 luglio 2012 : 18:39:43




|
| In pratica, nel disegno che hai appena aggiunto, il ss-break verrà molto più semplicemente riparato usando il filamento complementare dello stesso omologo, che è intatto. |
 |
|
|
JimmyCorgan
Nuovo Arrivato

5 Messaggi |
 Inserito il - 31 luglio 2012 : 19:24:55 Inserito il - 31 luglio 2012 : 19:24:55




|
Un pò più chiaro 
Ma una cosa. Nel caso G, i sistemi di riparazione che entrano in gioco nella regione etero-duplex sono sia endo che esonucleasi? E se si, allora potrebbero, per esempio, eliminare la sequenza E ri-sintetizzandola usando come stampo la sequenza e', facendo si che ci sia comunque una ricombinazione? |
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 31 luglio 2012 : 23:29:58 Inserito il - 31 luglio 2012 : 23:29:58





|
Citazione:
Messaggio inserito da roberta.s
Aggiungerei che, se si parla di ricombinazione omologa, c'è da precisare che essa è indotta molto più frequentemente da DSB che da single strand breaks, i quali sono solitamente riparati tramite NHEJ
Mi spiace contraddirti, ma quel che dici non è esatto, quando c'è errore a singolo filamento altri sono i sistemi di riparo. Il sistema NHEJ si attiva solo in seguito a danno al doppio filamento, cioè un DSB. Quelo che accade è che, in caso di DSB, la ricombinazione omologa non si verifica quasi mai, perchè si attiva subito il sistema NHEJ (anche il nome rende bene la sua funzione: non homologous end joining recombination). Durante la profase I della meiosi avrai svariati fenomeni di ricombinazione, mi pare di ricordare una cosa come 3 crossing over per cromosoma (di H. sapiens), per il restante tempo dell'individuo, la ricombinazione omologa rimane un evento molto raro.
Citazione:
Messaggio inserito da JimmyCorgan
Un pò più chiaro 
Ma una cosa. Nel caso G, i sistemi di riparazione che entrano in gioco nella regione etero-duplex sono sia endo che esonucleasi? E se si, allora potrebbero, per esempio, eliminare la sequenza E ri-sintetizzandola usando come stampo la sequenza e', facendo si che ci sia comunque una ricombinazione?
No, nella regione eteroduplex avrai soltanto casi di mismatch repair, non c'è DSB e quindi non verranno attivati ne NHEJ ne fenomeni di ricombinazione omologa, il cromosoma ha già dato per quest'ultima ^^ |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
roberta.s
Utente Junior


Città: Parigi

564 Messaggi |
 Inserito il - 01 agosto 2012 : 09:34:35 Inserito il - 01 agosto 2012 : 09:34:35




|
Citazione:
Messaggio inserito da zerhos
Citazione:
Il sistema NHEJ si attiva solo in seguito a danno al doppio filamento, cioè un DSB.
Hai ragione sulla terminologia, intendevo riparo sul filamento complementare dello stesso omologo, non so perché ho scritto NHEJ
[quote][i]Messaggio inserito da zerhos
[quote][i]Quelo che accade è che, in caso di DSB, la ricombinazione omologa non si verifica quasi mai, perchè si attiva subito il sistema NHEJ
Ma sta di fatto che sulla teoria non concordo. I DSBs vengono riparati con uno dei due meccanismi tra HR e NHEJ. I due meccanismi sono entrambi possibili, con la differenza che il NHEJ è più veloce ma error prone, mentre la ricombinazione omologa è più complessa ma error free. Inoltre, sono proprio i DSB a permettere agli omologhi, durante la meiosi, di creare quelle connessioni necessarie per la corretta segregazione degli stessi. Sono sempre i DSB, via riparo tramite ricombinazione omologa, a dare origine ai crossing over. Quindi non direi che in caso di DSB, la ricombinazione omologa non si verifica quasi mai.
|
 |
|
|
roberta.s
Utente Junior


Città: Parigi

564 Messaggi |
 Inserito il - 01 agosto 2012 : 09:41:53 Inserito il - 01 agosto 2012 : 09:41:53




|
Citazione:
Messaggio inserito da zerhos
[quote][i]Durante la profase I della meiosi avrai svariati fenomeni di ricombinazione, mi pare di ricordare una cosa come 3 crossing over per cromosoma (di H. sapiens), per il restante tempo dell'individuo, la ricombinazione omologa rimane un evento molto raro.
Certo, la ricombinazione omologa avviene soprattutto in meiosi.
Cito da wikipedia:
Homologous recombination is a type of genetic recombination in which nucleotide sequences are exchanged between two similar or identical molecules of DNA. It is most widely used by cells to accurately repair harmful breaks that occur on both strands of DNA, known as double-strand breaks. Homologous recombination also produces new combinations of DNA sequences during meiosis, the process by which eukaryotes make gamete cells, like sperm and egg cells in animals. These new combinations of DNA represent genetic variation in offspring, which in turn enables populations to adapt during the course of evolution.[1] Homologous recombination is also used in horizontal gene transfer to exchange genetic material between different strains and species of bacteria and viruses.
|
 |
|
|
marianna rosei
Nuovo Arrivato

1 Messaggi |
 Inserito il - 08 agosto 2012 : 00:24:53 Inserito il - 08 agosto 2012 : 00:24:53




|
| scusate come si fa a sapere quali proteine sono state finora sintetizzate con la tecnica del dna ricombinante? Un enzima di peso molecolare molto alto come la glutathione transferasi potrebbe essere clonata con questa tecnica. grazie |
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 09 agosto 2012 : 23:24:30 Inserito il - 09 agosto 2012 : 23:24:30





|
Citazione:
Messaggio inserito da marianna rosei
scusate come si fa a sapere quali proteine sono state finora sintetizzate con la tecnica del dna ricombinante? Un enzima di peso molecolare molto alto come la glutathione transferasi potrebbe essere clonata con questa tecnica. grazie
Cosa intendi, spiegati meglio. |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-18 MolecularLab.it |
 |
|
|








