Quanto č utile/interessante questa discussione:
| Autore |
Discussione |
|
|
bububebč
Utente Junior


Prov.: Na
Cittŕ: Napoli

255 Messaggi |
 Inserito il - 25 febbraio 2009 : 12:04:34 Inserito il - 25 febbraio 2009 : 12:04:34




|
 A TUTTI! A TUTTI!
Potreste seguire un attimo il mio ragionamento sulla maturazione dei miRNA per favore?so che per alcuni si tratta di una cosa banalissima,ma vi assicuro che non ho trovato nessuna spiegazione corretta del meccanismo(rispetto a come il mio prof. vuole esserlo spiegato)..voglio solo sapere se manca qualcosa.. dunque i pri-miRNA sono sintetizzi da RNA poli.II,(quanto sono lunghi?100nt?)ed hanno una struttura steam-loop con una piccola ansa sporgente nella parte superiore con appendici al 3' e al5'.Vengono processati da drosha e pasha,ottenendo pre-miRNA di 70-80nt che mancano delle appendici alle estremitŕ(?),da qui escono dal nucleo grazie all'NPC e arrivano nel citoplasma,dove la dicer,produce frammenti di 23nt detti miRNA(che forma hanno?come li processa l'enzima?in quali punti agisce e come modifica i pre-miRNA?).Si legano al complesso RISC,ed infine impediscono la traduzione del trascritto legandosi al 3'UTR per complementarietŕ imperfetta...č giusto? cosa manca in questo ragionamento?
Aspetto vostre risposte!Grazie mille a tutti  
|
|
|
|
|
GFPina
Moderatore
    

Cittŕ: Milano

8408 Messaggi |
 Inserito il - 25 febbraio 2009 : 18:02:27 Inserito il - 25 febbraio 2009 : 18:02:27





|
Innanzitutto grazie di aver riscritto il messaggio in Italiano, stavo rispondendo prima che si bloccasse internet e ci avevo messo un po' ad interpretare!
Comunque:
Citazione:
Messaggio inserito da bububebč
steam-loop
si scrive stem-loop che vuol dire "gambo-ansa" e riflette il tipo di struttura!
steam-loop vuol dire "vapore-ansa"
Tornando alla maturazione dei miRNA, questa mi sembra un'immagine abbastanza semplice ed esplicativa:
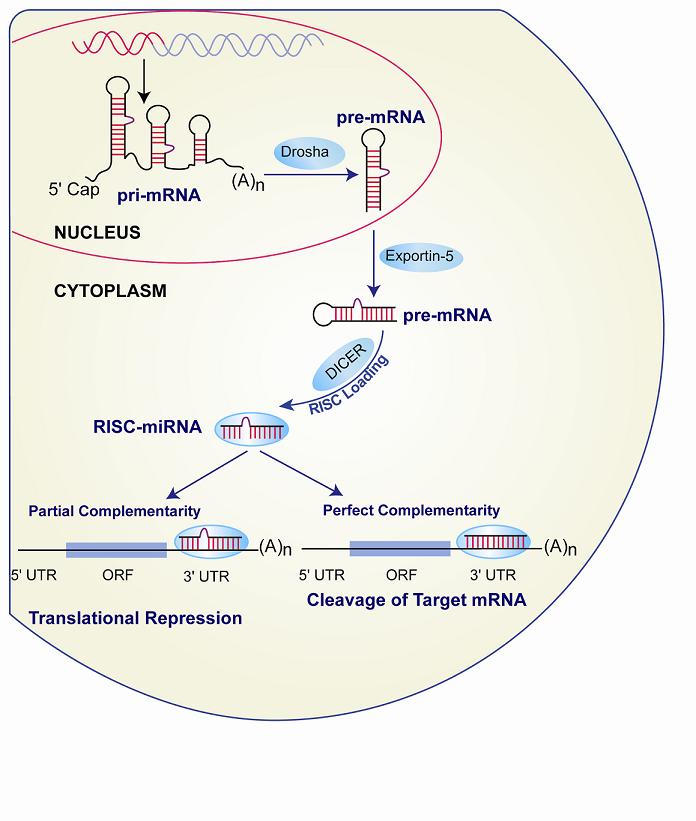
I pri-miRNA sono trascritti da parte della RNA polimerasi II (in realtŕ č stato scoperto che ci sono delle varianti in questo passaggio, ma in linea generale č cosě)
I geni per i miRNA possono essere singoli o raggruppati in cluster, che vengono trascritti tutti nello stesso pri-miRNA. Quindi i pri-miRNA possono essere formati solo dal precursore di un microRNA e presentano una singola struttura a forcina (stem-loop) oppure possono essere formati da precursori di vari miRNA e quindi presentare varie strutture a forcina (come puoi vedere dalla figura) ognuna delle quali rappresenta il precursore di un mRNA.
Per questo motivo la lunghezza dei pri-miRNA č molto variabile e puň essere anche di parecchie migliaia di basi.
In ogni caso i pri-miRNA contengono un cap al 5’ e una coda di polyA.
I pri-miRNA vengono processati da Drosha e Pasha (Partner of Drosha) e si ottengono i pre-miRNA, che sono singole struttura a forcina mancanti del cap e della coda di polyA.
I pre-miRNA vengono trasportati nel citoplasma attraverso i pori della membrana nucleare (NPC = nuclear pore complex) grazie ad un meccanismo di trasporto ad opera della Exportina 5.
Una volta nel citoplasma sono processati dall’enzima Dicer che taglia la parte ad ansa e si ottiene un miRNA a doppio filamento miRNA/miRNA* duplex, ogni filamento č di 20-22nt.
La maturazione del miRNA finisce con il legame al complesso RISC in cui viene selezionato solo un filamento, mentre l’altro complementare viene degradato.
In seguito il miRNA puň legarsi al suo mRNA target con due tipologie di legame:
Se il miRNA si lega in modo non complementare all’mRNA si ha l’inibizione della trascrizione, e questo č il meccanismo piů frequentemente osservato.
Il legame del miRNA all’RNA puň anche essere complementare e porta a degradazione dell’mRNA, questo meccanismo č presente soprattutto nelle piante, ma pare che alcuni mi-RNA agiscano in questo modo anche negli animali.
Qua trovi una figura piů completa: http://www.nature.com/nrmicro/journal/v4/n9/fig_tab/nrmicro1473_F1.html
Spero di aver scritto tutto tra le varie interruzioni di internet.
  |
 |
|
|
bububebč
Utente Junior


Prov.: Na
Cittŕ: Napoli

255 Messaggi |
 Inserito il - 25 febbraio 2009 : 20:33:46 Inserito il - 25 febbraio 2009 : 20:33:46




|
Che bella spiegazione!! sei stata chiarissima,finalmente ho capito anche la struttura specifica che all'esame bisogna illustrare..
E sorry per aver scritto in modo non chiaro prima .. ..
ti ringrazio tanto   ! ! |
 |
|
| |
Discussione |
|
|
|
Quanto č utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|




 A TUTTI!
A TUTTI!
