| Autore |
Discussione |
|
|
mollichina di pane
Utente Junior



121 Messaggi |
 Inserito il - 04 maggio 2009 : 16:27:09 Inserito il - 04 maggio 2009 : 16:27:09




|
Ciao a tutti!
Avrei una domanda...qualcuno di voi ha mai fatto una PCR su un prodotto già amplificato?
e se sì, come si fa? quanta PCR iniziale va utilizzata?
Spero tanto che qualcuno possa aiutarmi!!!
|
|
|
|
|
Aureus
Utente Junior


Prov.: Forlì-Cesena
Città: Gambettola

123 Messaggi |
 Inserito il - 04 maggio 2009 : 17:18:47 Inserito il - 04 maggio 2009 : 17:18:47





|
Ciao,
forse tua intenzione è di fare una PCR Nested...
...molto semplice, dalla tua I° PCR disegni dei primer interni "Inner", poi amplifichi con le tue condizioni ideali che avrai appurato con un semplice softwere tipo "Oligo"...
...molto spesso la PCR Nested consente di aumentare notevolmente la sensibilità e la specificità della PCR soprattutto nei casi in cui la presenza del targhet iniziale è molto bassa..
|
www.tlbspazio.it
Tecnici di Laboratorio Biomedico
Lorenzo Nardi |
 |
|
|
Aureus
Utente Junior


Prov.: Forlì-Cesena
Città: Gambettola

123 Messaggi |
 Inserito il - 04 maggio 2009 : 17:27:17 Inserito il - 04 maggio 2009 : 17:27:17





|
Come si fa...
...ovviamente le condizioni saranno quelle della prima PCR, tenendo conto che c'è più amplificato targhet...
Quanta PCR iniziale va utilizzata...
Io di solito utilizzo tutto il prodotto di PCR per fare la PCR secondaria..
...io di solito lo faccio per vedere il genoma di EBV HHV7 e HHV8 che in cellule infette non si trovano in enormi quantita..
...tu? Su cosa la fai?
Immagine:
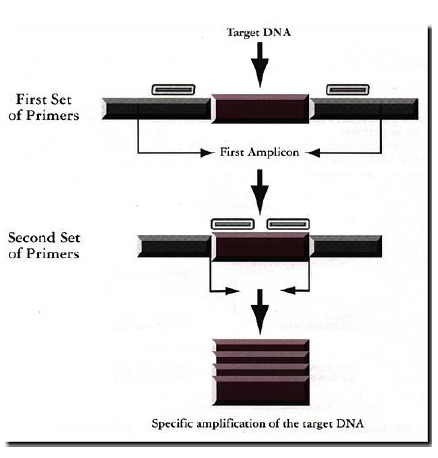
71,08 KB |
www.tlbspazio.it
Tecnici di Laboratorio Biomedico
Lorenzo Nardi |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 04 maggio 2009 : 18:09:07 Inserito il - 04 maggio 2009 : 18:09:07





|
Si a me è capitato di farle, non solo nested ma riamplificare con gli stessi primers il prodotto di PCR per svariati motivi, c'era anche una discussione in proposito.
Comunque dipende molto da quanto è il prodotto di PCR che ottieni, io per le nested ho sempre usato 1-5ul di prodotto di PCR diluito da 1:1000 a 1:10000.
EDIT: ecco la discussione in cui se ne parlava
http://www.molecularlab.it/forum/topic.asp?TOPIC_ID=9386
 |
 |
|
|
mollichina di pane
Utente Junior



121 Messaggi |
 Inserito il - 05 maggio 2009 : 12:33:44 Inserito il - 05 maggio 2009 : 12:33:44




|
Grazie ad entrambi...siete stati molto gentili.
Dunque...non avevo bisogno di fare una nested, ma i tuoi consigli, aureus, sono cmq ben accetti!!!
Intendevo fare una PCR e poi, sul prodotto riamplificare con gli stessi primer, come diceva GFPina.
Ad ogni modo...
...l'ho fatto ieri pomeriggio utilizzando 5uL della prima PCR ed oggi quando ho controllato sul gel, la mia banda c'era ed era ben forte, ma c'erano anche altre bande. In particolare una molto vicina a quella d'interesse.
Come posso spiegarmi questa cosa? (non si tratta di una contaminazione!ne sono certa)
Grazie |
 |
|
|
mak_steon
Utente Junior


138 Messaggi |
 Inserito il - 05 maggio 2009 : 16:36:26 Inserito il - 05 maggio 2009 : 16:36:26




|
I primer che utilizzi sono molto specifici? Magari col fatto che c'era una grande quantità di DNA si sono legati aspecificatamente.
Le bande secondarie sono molto intense?
Potrebbe essere opera di nucleasi inquinanti? (Possibilità molto remota lo so)
Una curiosità: dopo la prima PCR non vedevi proprio niente oppure una debole banda c'era?
Ciao ciao |
 |
|
|
mollichina di pane
Utente Junior



121 Messaggi |
 Inserito il - 07 maggio 2009 : 17:39:59 Inserito il - 07 maggio 2009 : 17:39:59




|
Allora...
...gli oligo che utilizzo sono specifici: mi servono per tirarmi giù l'inserto che dovrò clonare (posseggono, quindi, anche i siti per gli enzimi che ho scelto).
Le bande secondarie sono molto deboli tranne una, molto vicina come peso al mio amplicone.
Le nucleasi le escluderei!
La prima PCR che ho fatto aveva solo la mia banda d'interesse e per giunta molto molto forte; nessuna banda aspecifica, nemmeno debolissima.
Chissà che roba sono quelle bande?!
Ciao e grazie |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 07 maggio 2009 : 19:54:22 Inserito il - 07 maggio 2009 : 19:54:22





|
Possono essere degli aspecifici! Se la tua PCR era già buona prima e avevi una bella banda 5ul della prima PCR (in non so quanto) sono veramente tanti, è normale che ti saltino fuori anche cose aspecifiche. Potresti aver avuto qualcosa di aspecifico anche nella prima PCR che non vedevi ma riamplificando ti salta fuori.
Prova a fare la seconda PCR partendo da meno templato.
E poi al massimo estrarrai la tua banda da gel.
Ma una cosa mi sfugge, ma se la banda era già bella nella prima PCR perchè fai anche la seconda?
Forse che la prima era con primers senza siti di restrizione e la seconda con gli stessi primers ma con i siti di restrizione? |
 |
|
|
mollichina di pane
Utente Junior



121 Messaggi |
 Inserito il - 12 maggio 2009 : 16:51:43 Inserito il - 12 maggio 2009 : 16:51:43




|
Grazie del consiglio...se dovesse ricapitare lo seguirò!
La cosa è un po' complicata...dovevo inserire una sequenza in un vettore prendendola da un altro vettore.
Purtroppo questo secondo vettore mi è finito e non abbiamo soldi per ricomprarlo.
La prima PCR che ho fatto è andata bene, ma poi nei vari passaggi fino alla restrizione, il mio frammento è scomparso (il kit con cui eluisco da gel ha rese piuttosto basse!).
Alla fine mi sono detta:" ok...ho ancora 5 uL di PCR...cosa posso farci?" e così...
Ecco tutto. Avevo anche pensato di fare una PCR inversa inserendo la sequenza che mi serviva direttamente sul vettore, ma inserzioni così grandi non le ho mai provate...dovrei mettere a punto un protocollo. Ho optato per la cosa che mi sembrava più fattibile nell'immediato.
Grazie ancora!!!  |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 12 maggio 2009 : 18:26:37 Inserito il - 12 maggio 2009 : 18:26:37





|
Ah ecco svelato il mistero! 
Spero che il clonaggio alla fine sia venuto bene!
  |
 |
|
|
miky76
Utente Junior


126 Messaggi |
 Inserito il - 13 maggio 2009 : 01:32:43 Inserito il - 13 maggio 2009 : 01:32:43




|
La cosa è un po' complicata...dovevo inserire una sequenza in un vettore prendendola da un altro vettore.
Purtroppo questo secondo vettore mi è finito e non abbiamo soldi per ricomprarlo.
ciao
anche io spero che il tuo clonaggio sia ok
ma domanda,,, forse stupida...
ma perche' prima di finire il vettore non l'hai riamplificato?
anche se e' un vettore commericiale
si puo' amplificare tranquillamente

|
 |
|
|
mollichina di pane
Utente Junior



121 Messaggi |
 Inserito il - 13 maggio 2009 : 12:03:48 Inserito il - 13 maggio 2009 : 12:03:48




|
Spero anch'io che sia venuto bene...nel senso che sto screenando dei cloni positivi alla colony-PCR, sui quali faro altri controlli domani. E poi...solo il sequenziatore mi dirà se è venuto o meno!!!
"ma perche' prima di finire il vettore non l'hai riamplificato?
anche se e' un vettore commericiale"
Perchè era un vettore linearizzato presente in un kit in cui mancava praticamente tutto  ...alla fine ho optato per la PCR con tanto di risciacquo di provetta contenente il vettore!!! ...alla fine ho optato per la PCR con tanto di risciacquo di provetta contenente il vettore!!!
grazie ancora a tutti!
 |
 |
|
| |
Discussione |
|







