| Autore |
Discussione |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 16 settembre 2009 : 13:22:33 Inserito il - 16 settembre 2009 : 13:22:33




|
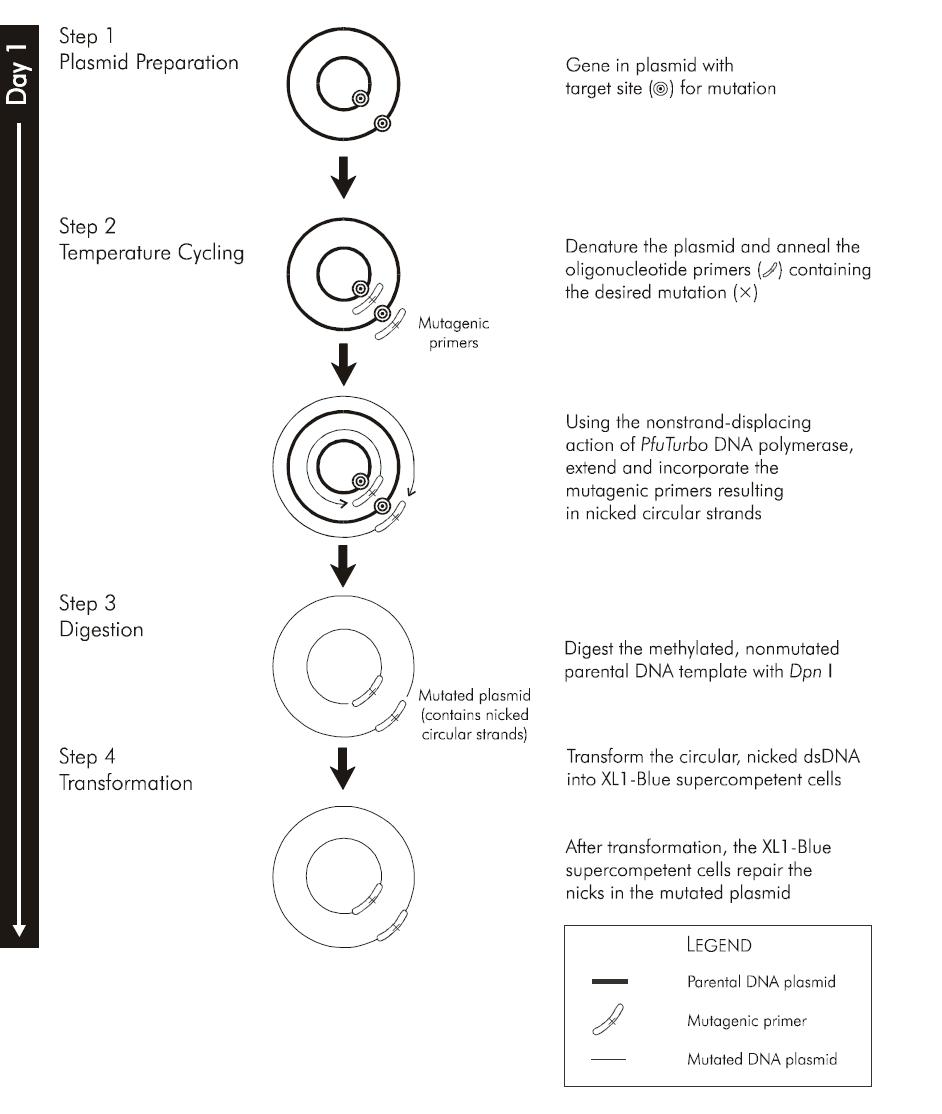
Un dubbio atroce:
Ma il DNA circolare (batterico, plasmidico) sottoposto a denaturazione si separa come un DNA lineare in 2 filamenti o rimane ancorato in un punto? (Modello anelli delle olimpiadi per intenderci)
Perchè altrimenti non ho capito bene st'immagine?
Dopo aver amplificato con la pfu ottengo 2 plasmidi separati, ognuno con un elica parentale ed una di neosintesi o una struttura comprendente ben 4 eliche?(Ammesso che non sia altamente instabile).
Grazie
Immagine:
94,61 KB
|
|
|
|
|
Neuroscience
Utente
 

659 Messaggi |
 Inserito il - 16 settembre 2009 : 14:06:03 Inserito il - 16 settembre 2009 : 14:06:03




|

Non so se ho capito cosa vuoi sapere, però l'immagine dice che da ogni filamento di DNA parentale si sintetizzano due nuovi vettori circolari a doppio filamento di cui uno è parentale e l'altro è neosintetizzato.
Ovviamente quello di neosintesi non è perfettamente chiuso e quindi può abbandonare il DNA parentale senza fare i cerchi delle olimpiadi.
Non si formano strutture di DNA a 4 filamenti e la figura è solo rappresentativa, in realtà i due filamenti di DNA parentali sono nettamente divisi lontani, ed ognuno di questi darà luogo alla neosintesi dell'altro filamento mancante.
Alla fine dei x cicli ottieni una predominanza di DNA sintetizzato dalla Pfu, magari nikkato, ed una piccola porzione di DNA plasmidico mixato tra la neositesi da pfu ed il DNA parentale.
 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 16 settembre 2009 : 14:18:55 Inserito il - 16 settembre 2009 : 14:18:55





|
Premesso quello che ha detto Neuroscience, tieni comunque presente che c'è differenza tra il DNA plasmidico sottoposto a PCR e la replicazione del DNA genomico dei batteri. (visto che hai nominato anche quello!)
Durante la replicazione del DNA genomico questo non viene denaturato completamente (estattamente come per gli eucarioti) e si forma una bolla di replicazione, questo dà origine alla tipica forma a "teta"
 |
 |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 16 settembre 2009 : 15:40:27 Inserito il - 16 settembre 2009 : 15:40:27




|
Grazie mille ragazzi sapevo di poter contare su di voi.
Per completezza vi lascio questo link, così magari potete darci un'occhiata. La storia del Quickgen kit adesso mi è chiara, per quanto riguarda l'idea della denaturazione del dna a doppia elica circolare, tutti i dubbi mi sono nati da questa lettura trovata casualmente.
link http://www.notahelix.com/not_a_helix_files/index-tn.htm
 |
 |
|
| |
Discussione |
|







