| Autore |
Discussione |
|
|
giufalab
Nuovo Arrivato

15 Messaggi |
 Inserito il - 22 ottobre 2009 : 15:01:53 Inserito il - 22 ottobre 2009 : 15:01:53




|
Ciao a tutti!! sono un paio di mesi che mi sto esaurendo sulla genotipizzazione di topi laron.
Dovrei avere bande differenti a seconda del genotipo:
Wild Type = 1 banda a 250
Hetero = 2 bande
Laron = 1 banda a 350
utilizzo 3 primer già utilizzati in bibliografia.
non sembrerebbe niente di complicato se non fosse che le bande a volte ci sono e a volte.
mi è capitato molte volte di ripetere la pcr nelle stesse condizioni, con le stesse quantità di dna e al 2/3 tentativo finalmente avevo le mie bande.
Dato che i risultati dovrebbero venire alla prima botta, qualcuno ha qualche idea??
sapete se l'utilizzo di 3 primer può dare problemi?
grazie mille per la risposta.
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 22 ottobre 2009 : 16:23:16 Inserito il - 22 ottobre 2009 : 16:23:16





|
Io non ho esperienza con questi topi, ma ho utilizzato varie PCR per altri topi, sia a 3 che a 4 primers e le mie ora funzionano bene, ma per alcune ho avuto problemi.
Mi è capitao però una volta che per fare un genotipo di alcuni topi utilizzavo i primers consigliati dalla Jackson che forniva i topi (primers disegnati dal creatore di quei topi transgenici) e funzionava malissimo, avevo un sacco di bande aspecifiche, ho ridisegnato io i primers e la PCR funzionava alla perfezione.
Per altri topi invece in cui devo valutare se c'è delezione di un pezzo di DNA ho un sistema a 3 primer con sequenze prese dalla letteratura, ho dovuto variare un po' le condizioni di PCR per trovare le condizioni ottimali perché i risultati erano un po' altalenanti, ma con un po' di prove ho risolto il problema.
Sai ogni PCR è un caso a sé, quindi non si può mai dire, puoi provare a cambiare le condizioni e se non risolvi il problema provare a ridisegnare i primer. Purtroppo i primers presi dalla letteratura non sempre funzionano! 
Ah dimenticavo, hai provato anche a fare la PCR in provette separate? Una per il WT e una per il Laron? In questo modo puoi valutare se le PCR indipendenti vengono e nel caso qual'è il primer che da dei problemi. Puoi provare a settare le condizioni prima in PCR separate e poi mettere tutto assieme nella stessa PCR.
Insomma, l'unica è fare delle prove.
 |
 |
|
|
giufalab
Nuovo Arrivato

15 Messaggi |
 Inserito il - 22 ottobre 2009 : 16:44:40 Inserito il - 22 ottobre 2009 : 16:44:40




|
i primer li devo usare in contemporanea per amplificare il gene di mio interesse.
ora provo a variare un pò le condizioni e spero che mi venga bene.
grazie |
 |
|
|
Soshi Fujiwara
Nuovo Arrivato


60 Messaggi |
 Inserito il - 22 ottobre 2009 : 17:53:02 Inserito il - 22 ottobre 2009 : 17:53:02




|
Quoto GFPina per quanto riguarda il fatto che spesso i primer segnalati negli articoli non funzionano a dovere e, se hai a disposizione le sequenze, è meglio se li ridisegni.
Riguardo invece al cambiare le condizioni di PCR io ultimamente ho fatto un pò di test per capire come cambiavano i prodotti modificando le concentrazioni dei reagenti a parità di programma di amplificazione e ho notato che il rapporto MgCl2\dNTPs è fondamentale, anche minime variazioni influenzano il risultato più che nel caso della variazione del solo MgCl2.
Il rapporto MgCl2\dNTPs:
- se diminuisce; diminuisce anche la specificità, ovvero ottengo più polimorfismi
- se aumenta; aumenta anche la specificità, ovvero ottengo meno polimorfismi
il tutto a parità di ciclo di amplificazione.
Solitamente io li uso in rapporto 10:1 quando ho primer molto specifici e il risultato è ottimo (es. 1,7mM:0,17mM). Una mia collega per fare dei RAPD e cercare polimorfismi usa 1,5mM:0,22mM e il numero di bande è almeno 10 volte maggiore. Se il rapporto scende sotto gli 8,5 o supera gli 11,8 c'è un crollo e i gel sono vuoti.
Spero di essere stato d'aiuto in qualche modo... |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 22 ottobre 2009 : 18:37:01 Inserito il - 22 ottobre 2009 : 18:37:01




|
Guarda, ho avuto lo stesso problema con una PCR a 3 primer.
Concordo con il suggerimento di GFPina di fare 2 PCR separate (una con il primer comune e quello wt e l'altra col primer comune e il mutante). Nel mio caso aveva funzionato, vale la pena provare. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
giufalab
Nuovo Arrivato

15 Messaggi |
 Inserito il - 22 ottobre 2009 : 19:47:37 Inserito il - 22 ottobre 2009 : 19:47:37




|
i primer mi servono tutti e 3 perchè sono tutti sullo stesso gene.
2 sono i classici primer il 3 mi serve per la mutazione all'interno del gene, senza sarebbero tutti wild type... |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 22 ottobre 2009 : 21:00:01 Inserito il - 22 ottobre 2009 : 21:00:01




|
Scusa, ma se hai un set di 3 primer, ne avrai uno che viene usato sia da wt che mutato e gli altri che sono usati solo in uno dei due, no?
Se è così puoi fare 2 PCR come ti abbiamo detto, usando una volta il primer comune e quello wt e l'altra volta il comune e quello per il mutato.
Oppure la situazione è differente? (ma non mi viene in mente alcuna altra soluzione) |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 22 ottobre 2009 : 21:12:02 Inserito il - 22 ottobre 2009 : 21:12:02





|
Citazione:
Messaggio inserito da giufalab
i primer mi servono tutti e 3 perchè sono tutti sullo stesso gene.
2 sono i classici primer il 3 mi serve per la mutazione all'interno del gene, senza sarebbero tutti wild type...
Scusa presumo che tu debba sapere come è stato fatto il tuo topo!
Se non lo sai ti consiglio di leggere l'articolo originale: A mammalian model for Laron syndrome produced by targeted disruption of the mouse growth hormone receptor/binding protein gene (the Laron mouse)
in ogni caso è un banalissimo classico KO ed è stato fatto inserendo una cassetta in modo da creare una delezione del gene, in questo modo:
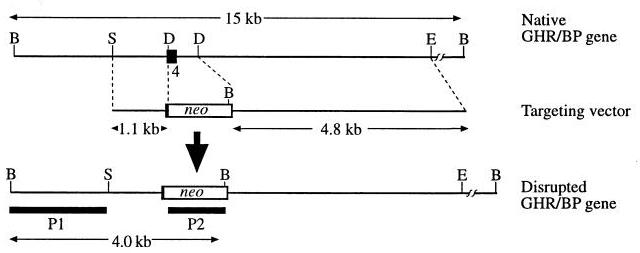
quelle segnate in figura sono le sonde utilizzate per il Southern nell'articolo. Ma in ogni caso se hai un sistema a 3 primers che ti da:
Citazione:
Wild Type = 1 banda a 250
Hetero = 2 bande
Laron = 1 banda a 350
ovviamente avrai:
- un primer esterno alla zona deleta che si lega sia al WT che al Laron
- un primer nella zona deleta nel Laron che quindi si lega solo al WT
- un primer nella cassetta inserita nel Laron che quindi si lega solo al Laron
in questo modo hai la presenza delle 2 bande diverse.
Credimi questo è l'unico modo perché il sistema possa funzionare!!! 
Altrimenti non avrebbe senso.
Quindi come ti abbiamo detto sia io che chick80 puoi fare le due PCR separate!
L'unica cosa che non so, non conoscendo le sequenze dei tuoi primers è se il primer che lega entrambi è forward o reverse (e quindi gli altri 2 saranno reverse nel primo caso o forward nel secondo), ma ci vuole poco a capirlo nel caso tu non lo sappia, basta fare un blast con la sequenza del gene.
 |
 |
|
|
Gst
Utente Junior


Prov.: Roma
Città: Ariccia

143 Messaggi |
 Inserito il - 25 ottobre 2009 : 14:21:06 Inserito il - 25 ottobre 2009 : 14:21:06




|
Anzichè fare una PCR unica con 3 primers, in caso di problemi spesso basta fare 2 PCR separate SEMPRE: utilizzando sugli stessi campioni le 2 diverse coppie di primers separatamente. In questo modo si può ottimizzare la PCR su temperature di melting + adatte alle singole coppie di primers.
Nono pensate?
Anche se apisco che fare una sola PCR a 3 primers è più veloce e meno dispendioso. |
Ascoltami con gli occhi... |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 25 ottobre 2009 : 15:31:46 Inserito il - 25 ottobre 2009 : 15:31:46





|
| Beh si concordo sicuramente è più dispendioso se hai un sistema a 3 (o anche 4 primers) per il genotipo da utilizzare nella stessa PCR che funziona bene, conviene di sicuro. |
 |
|
|
giufalab
Nuovo Arrivato

15 Messaggi |
 Inserito il - 25 ottobre 2009 : 16:08:46 Inserito il - 25 ottobre 2009 : 16:08:46




|
Grazie a tutti per i consigli.
Il problema potrebbe essere anche di natura strumentale.
il nostro ternociclizzatore fa le bizze e il tecnico ha detto che potrebbe non fare bene il suo lavoro 
e io che cercavo mille soluzioni....
in settimana provo un'altra macchina per vedere cosa succede, nel caso vada male provo a fare pcr con primer separati
|
 |
|
|
giufalab
Nuovo Arrivato

15 Messaggi |
 Inserito il - 28 ottobre 2009 : 18:57:31 Inserito il - 28 ottobre 2009 : 18:57:31




|
Dopo tanti ragionamenti, ipotesi su ipotesi, teorie e prove sono arrivata alla conclusione che il mio ciclizzatore è passato a miglior vita 
ossia la piastra riscalda in alcune parti di più e altre meno quindi la reazione a volte va bene altre no.
grazie a tutti per l'aiuto.
Giulia |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 29 ottobre 2009 : 00:50:56 Inserito il - 29 ottobre 2009 : 00:50:56





|
Beh buono a sapersi che hai trovato qual'è il problema!
 |
 |
|
| |
Discussione |
|




