| Autore |
Discussione |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
 Inserito il - 23 febbraio 2010 : 17:33:57 Inserito il - 23 febbraio 2010 : 17:33:57




|
Un po' per problemi con le Real Time, un po' perchè abbiamo altre RT in prova stiamo testando le diverse RT. Pensavamo di fare semplicemente un rapporto quantità/prezzo, invece ci siamo accorti che con RT diverse otteniamo risultati diversi alla Real Time PCR  . .
Ad esempio questo è uno stesso campione (Rna) Retrotrascritto con 2 RT di due aziende diverse e guardate i risultati della melting:
Immagine:
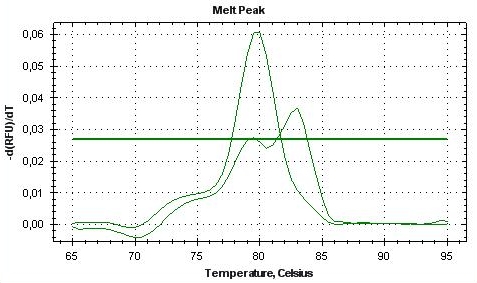
79,52 KB
Qualcuno mi sa spiegare il perchè?
In un altro test poi siamo andati a vedere la presenza di un gene che
doveva essere espresso molto in una linea cellulare e poco in un altra. Nonostante questa volta le melting siano venute bene, 2 RT indicano una differenza di 2 volte, un'altra RT di 100 volte! Non ci sto capendo niente...
Qualcuno può darmi qualche chiarimento?
|
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 23 febbraio 2010 : 18:14:55 Inserito il - 23 febbraio 2010 : 18:14:55





|
Dovresti darci maggiori info, tipo il metodo di RT utilizzato sopratutto se usa oligo-dT, random primers... e in realtà anche il protocollo è importante. Altra cosa importante è il gene che poi vuoi analizzare con la Real-time, cioè lunghezza dell'mRNA e dove sono posizionati i primers che utilizzi per la PCR... insomma i fattori da valutare sono veramente molti.
Un esempio banale: mRNA molto lungo i tuoi primers sono all'inizio del gene e retrotrascrivi con oligodT --> sarà difficile che l'mRNA venga retrotrascritto fino in fondo e che quindi tu abbia quella sequenza su cui fai la PCR. |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
 Inserito il - 03 marzo 2010 : 16:25:22 Inserito il - 03 marzo 2010 : 16:25:22




|
Purtroppo in questo giorni non ho avuto "tempo libero" e "dati sottomano" contemporaneamente, quindi ho rimandato un po'...
Oggi però ho avuto un po' di tempo e mi sono messo a ri-analizzare i dati, protocolli e caratteristiche dei vari kit, eccoli:
RT: Improm Promega| QuantiTect Qiagen | GoScript Promega
Melting: 2 picchi | 1 picco | 2 picchi
Primer cDNA: random,nostri | random, del kit | random, del kit
extra: / |elimina genomic DNA| /
amplifico diversi geni, in questo caso specifico ccl2, i primer sono sull'esone 2 e sull'esone 3.
Mentre cercavo tutti questi dati (questi primer non li ho disegnati io) mi sono accorto che l'amplificato su mRna dà un prodotto di circa 100 nt, mentre su genomico solo di 400 nt(!!!!quindi può essere amplificato!!!!!!), e tenendo conto che l'unica RT che dà questo "misterioso aspecifico" è quella che elimina il genomico...forse ho trovato la soluzione al mio problema!!! Evidentemente non estraggo correttamente l'Rna e mi porto un po' di genomico dietro! Che ne dici? Comunque Grazie Mille GFPina!!! |
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 03 marzo 2010 : 17:10:55 Inserito il - 03 marzo 2010 : 17:10:55





|
Sì decisamente, a metà del tuo discorso mi sono resa conto che al 99,9% il motivo è proprio quello a cui sei giunto tu, ovvero amplifichi il genomico!
Una cosa avevo notato subito dal tuo grafico con i picchi delle Tm, il secondo picco ("aspecifico") ha una Tm superiore a quella del picco "specifico", questo ti indica che hai un amplificato di dimensioni maggiori, quindi è decisamente consistente con il fatto che tu abbia amplificato del genomico. Se invece ci fosse un aspecifico dovuto ad esempio da dimeri di primers osserveresti un picco ad una Tm inferiore.
Potresti anche andarti a calcolare la Tm degli amplificati (da cDNA e da gDNA) per avere un'ulteriore conferma (con programmi che probabilmente conosci meglio di me!), ma in linea di massima direi che il tuo problema è quello.
In ogni caso il consiglio è sempre:
1° cercare di disegnare almeno uno dei due primer per la RT-PCR che si appai solo al cDNA e non al DNA
2° trattare l'RNA con DNasi
Io a dire la verità cerco di disegnare sempre primers che non amplifichino in alcun caso il genomico e evito di trattare con DNasi, anche se comunque il fatto di non trattare con DNasi rischia di darti una lettura un po' "falsata" allo spettrofotometro nel caso tu abbia nello stesso campione sia RNA che DNA.
Comunque se hai primers che di sicuro amplificano anche genomico il trattamento con DNasi lo devi necessariamente fare.
A questo punto ti consiglierei per continuare le tue prove con i diversi kit di trattare prima l'RNA con DNasi (nel caso 1 e 3) e ripetere la Real-time.
Un'altra cosa che faccio io (non assolutamente necessaria, ma io preferisco farla) è testare i primers prima con una PCR end-point. In questo caso avresti probabilmente visto le 2 bande a 100 e 400bp.
Anche dopo aver fatto la real-time comunque nel caso tu abbia risultati "strani" puoi sempre seminare i prodotti su gel di agarosio, ci vuole veramente poco e ti può dare un'indicazione di cosa sia successo.
 |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
 Inserito il - 16 marzo 2010 : 09:37:01 Inserito il - 16 marzo 2010 : 09:37:01




|
Ho ripetuto la real time su quei campioni, usando il kit che mi dava l'aspecifico e trattando una parte dell'Rna con DNasi. Il risultato si commenta da solo (anche se il picco non è forte come l'altra volta).
Immagine:
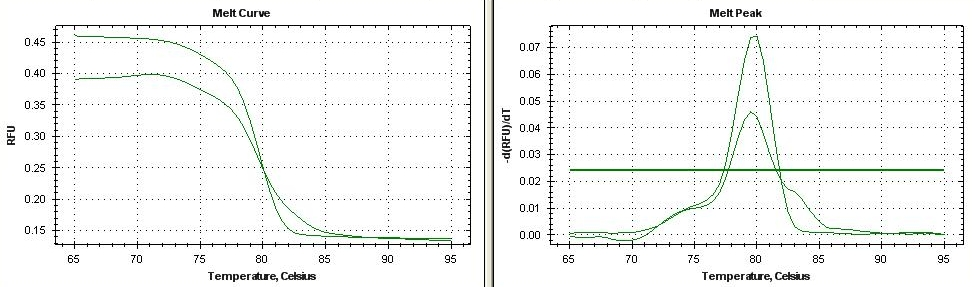
146,8 KB
Ho cercato di usare qualche tool per la temp di melting, ma non mi convincevano (chiedevano troppi dati che non sapevo). Tu me ne consigli qualcuno?
Comunque sembro aver risolto, no?! |
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 16 marzo 2010 : 16:38:14 Inserito il - 16 marzo 2010 : 16:38:14





|
Beh non so se ho capito giusto il grafico (guarda che si possono anche utilizzare differenti colori per le linee  ) una curva (quella con picco maggiore) è il retrotrascritto col kit che elimina l'RNA e il secondo (più basso) quello con trattato con DNasi? ) una curva (quella con picco maggiore) è il retrotrascritto col kit che elimina l'RNA e il secondo (più basso) quello con trattato con DNasi?
Comunque sia:
Citazione:
Comunque sembro aver risolto, no?!
mi sembra proprio di sì! Anche se decisamente è meglio il primo caso, nel secondo non hai più il doppio picco, ma comunque una gobba sulla curva nello stesso punto dove avevi il secondo picco.
Citazione:
Ho cercato di usare qualche tool per la temp di melting, ma non mi convincevano (chiedevano troppi dati che non sapevo). Tu me ne consigli qualcuno?
Ehmmmm speravo nel contrario, comunque non ne conosco molti a parte quelli segnalati su MFold: http://mfold.bioinfo.rpi.edu/cgi-bin/dna-form1.cgi
non sono esperta però. |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
 Inserito il - 07 aprile 2010 : 10:23:16 Inserito il - 07 aprile 2010 : 10:23:16




|
Mmm...non mi ero accorto che avevi risposto...
Allora, ecco i picchi con colori differenti (non lo sapevo si potesse fare... ) )
Immagine:
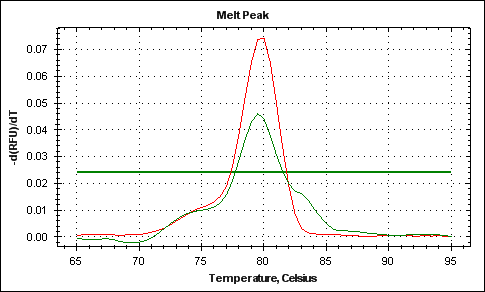
10,7 KB
Quello rosso è con la DNasi, quindi sparisce la gobbetta del doppio picco.
|
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 07 aprile 2010 : 10:31:55 Inserito il - 07 aprile 2010 : 10:31:55





|
Citazione:
Messaggio inserito da domi84
Mmm...non mi ero accorto che avevi risposto...

Citazione:
Messaggio inserito da domi84
Allora, ecco i picchi con colori differenti (non lo sapevo si potesse fare... ) )
Eheh si impara sempre qualcosa di nuovo! 
Citazione:
Messaggio inserito da domi84
Quello rosso è con la DNasi, quindi sparisce la gobbetta del doppio picco.
Ok ora mi è chiaro... problema risolto comunque!  |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
|
| |
Discussione |
|






