Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
peptina85
Nuovo Arrivato

3 Messaggi |
 Inserito il - 18 gennaio 2011 : 21:29:05 Inserito il - 18 gennaio 2011 : 21:29:05




|
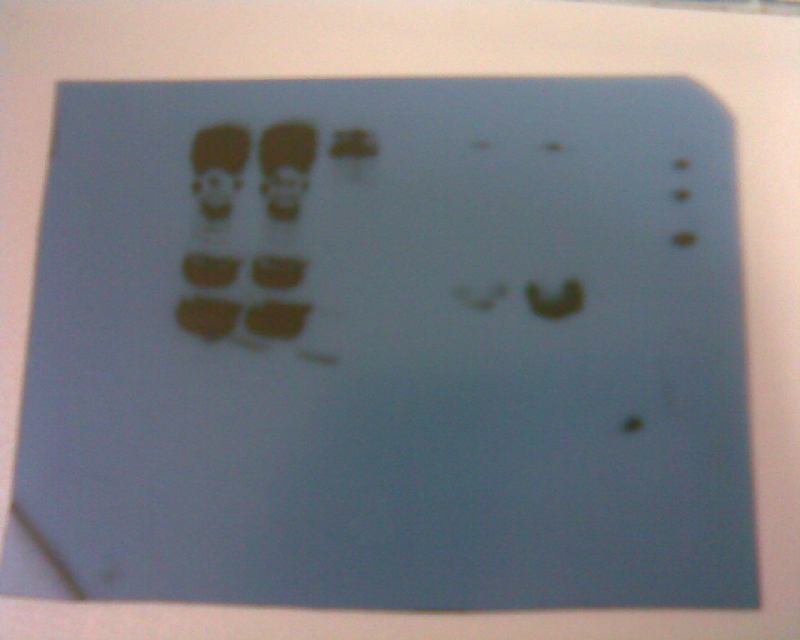
Ciao ragazzi..avrei bisogno del vostro aiuto...vorrei sapere come posso interpretare una lastra autoradiografica. Sono alle prese con esperimenti di coimmunoprecipitazione con i quali dovrei verificare l'interazione tra due proteine. In sintesi: i lisati, gli immunoprecipitati e i controlli vengono separati con SDS-PAGE e le proteine vengono poi trasferite dal gel alla membrana che poi verrà incubata, prima con un anticorpo primario e successivamente con il secondario. Si procede poi con la rivelazione ottenendo una lastra di questo tipo:
Immagine:
226,22 KB
In generale, vorrei capire i metodi che si usano per poter interpretare le lastre in modo che io possa identificarne i tratti più importanti.
Purtroppo non ho trovato alcun tipo di spiegazione nè su internet nè sui libri di testo che ho a disposizione.
Vi ringrazio.
|
|
|
|
|
Iside
Utente Attivo
  

Città: Napoli

1375 Messaggi |
 Inserito il - 18 gennaio 2011 : 21:49:59 Inserito il - 18 gennaio 2011 : 21:49:59





|
ma che cosa vuol dire "interpretare le lastre nei tratti più importanti"?
ogni lastra è un esperimento diverso quindi non è possibile uniformare i risultati. In generale, cerchi di vedere se la banda che hai rivelato è ad un PM corrispondente a quello suggerito dal data sheet dell'anticorpo, vedi se tra ctrl e trattati ci sono modulazioni di bande o shift di PM, cerchi di migliorare le incubazioni ed i lavaggi in base al background che ottieni...non so se sono questi i suggerimenti che cercavi... |
...we're just two lost souls swimming in a fishbawl...year after year...
|
 |
|
|
peptina85
Nuovo Arrivato

3 Messaggi |
 Inserito il - 19 gennaio 2011 : 21:56:30 Inserito il - 19 gennaio 2011 : 21:56:30




|
Citazione:
Messaggio inserito da peptina85
Ciao ragazzi..avrei bisogno del vostro aiuto...vorrei sapere come posso interpretare una lastra autoradiografica. Sono alle prese con esperimenti di coimmunoprecipitazione con i quali dovrei verificare l'interazione tra due proteine. In sintesi: i lisati, gli immunoprecipitati e i controlli vengono separati con SDS-PAGE e le proteine vengono poi trasferite dal gel alla membrana che poi verrà incubata, prima con un anticorpo primario e successivamente con il secondario. Si procede poi con la rivelazione ottenendo una lastra di questo tipo:
Immagine:

226,22 KB
In generale, vorrei capire i metodi che si usano per poter interpretare le lastre in modo che io possa identificarne i tratti più importanti.
Purtroppo non ho trovato alcun tipo di spiegazione nè su internet nè sui libri di testo che ho a disposizione.
Vi ringrazio.
Immagine:
6,77 KB
Ciao Iside...grazie per la risposta tempestiva . .
Quanto hai detto, a livello generale, è la conferma di quello che già sapevo.
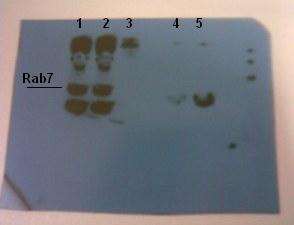
Ora provo a spiegare in modo più preciso qual è il mio dubbio: le proteine che devono interagire sono Rab7 e vimentina. I campioni che sono stati caricati su gel sono dei lisati ottenuti da precedenti trasfezioni effettuate in cellule Hela con due plasmidi ricombinanti contenenti due sequenze codificanti differenti, infatti sono sequenze che codificano per porzioni diverse della stessa proteina, la vimentina; un controllo, ossia un non trasfettato (NT), e gli immuno-precipitati (IP) sempre delle due porzioni della stessa proteina. Ho incubato la membrana con un anti-Rab7 prodotto in rabbit, il secondario ovviamente è un anti-rabbit e ho ottenuto quella lastra.
Nella lane 1 e nella lane 2 abbiamo caricato i lisati e come vediamo le bande presenti sono uguali,nella lane 3 abbiamo caricato il NT e a seguire, nella lane 4 e nella lane 5 abbiamo caricato gli IP e qui vediamo una differenza nel segnale, infatti è più intenso in corrispodenza di una delle due porzioni della proteina.
Inoltre la banda corrispondente al PM della mia proteina(Rab7)la ritrovo in quattro campioni e non la ritrovo nel NT. Perchè? Come la posso discutere questa lastra? |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 22 gennaio 2011 : 18:13:27 Inserito il - 22 gennaio 2011 : 18:13:27




|
Questo è un classico esperimento di delezione funzionale di una proteina, cioè ti dice quali domini all'interno di una proteina sono coinvolti in una sua particolare funzione (in questo caso quale porzione o dominio della vimentina è implicato nell'interazione con Rab7). Quindi hai immunoprecipitato con un anticorpo anti-dominio vimentina (immagino fossero taggati con flag o altro) e poi hai rivelato la presenza di rab7 con un ab specifico per rab7. Questo esperimento in soldoni ti dice che la porzione precipitata in 5 è quella "maggiormente" coinvolta nella interazione Vimentina-Rab7 (qualcsa si vede infatti anche nella lane 4).
Veniamo ai controlli: in lane 3 hai un nn trasfettato che presumo abbiate precipitato con lo stesso ab usato per gli ip: se cosi', esso ti serve da controllo negativo, cioè esclude un'eventuale interazione aspecifica anticorpo immunoprecipitante/rab7 che, qualora ci fosse, ti darebbe dei falsi positivi.
Le lane 1 e 2 corrispondono ai lisati totali, gli input che tu usi nella immunprecipitazione. Ti confermano la presenza di rab7 nel sistema (cellule hela) che tu usi. E' un controllo molto importante, perchè se non hai la proteina qui ovviamente non la tirerai giu' manco con la ip (ti esclude quindi i falsi negativi).
3 piccole annotazioni:
1) forse come controllo nella lane 3 sarebbe piu' corretto mettere un trasfettato con plasmide nudo, cioé senza inserto (senza le parti della proteina vimentina). Non ti cambia nulla da un punto di vista sperimentale, ma personalmente lo vedo piu' corretto da un punto di vista formale.
2)dici che la rab7 ti corre in tutte le lanes alla stessa altezza, ma a me nelle lane 4 e 5 mi paiono un po' piu' basse, non so dirti perchè, forse è solo un effetto della lastra (magari è storta) oppure dipende dalla corsa del gel... boh
3) andrebbe inclusa anche la rivelazione della proteina vimentina (o meglio delle porzioni di essa). Sei sicura che le cellule esprimano quantità equivalenti delle due porzioni? Magari il dominio in lane 4 è meno stabile, viene degradato piu' facilmente e di conseguenza ti precipita meno rab7. Oppure hai immunoprecipitato meno in sample rispetto all'altro... Io lo farei |
 |
 |
|
|
peptina85
Nuovo Arrivato

3 Messaggi |
 Inserito il - 23 gennaio 2011 : 21:05:12 Inserito il - 23 gennaio 2011 : 21:05:12




|
Citazione:
Messaggio inserito da SpemannOrganizer
Questo è un classico esperimento di delezione funzionale di una proteina, cioè ti dice quali domini all'interno di una proteina sono coinvolti in una sua particolare funzione (in questo caso quale porzione o dominio della vimentina è implicato nell'interazione con Rab7). Quindi hai immunoprecipitato con un anticorpo anti-dominio vimentina (immagino fossero taggati con flag o altro) e poi hai rivelato la presenza di rab7 con un ab specifico per rab7. Questo esperimento in soldoni ti dice che la porzione precipitata in 5 è quella "maggiormente" coinvolta nella interazione Vimentina-Rab7 (qualcsa si vede infatti anche nella lane 4).
Veniamo ai controlli: in lane 3 hai un nn trasfettato che presumo abbiate precipitato con lo stesso ab usato per gli ip: se cosi', esso ti serve da controllo negativo, cioè esclude un'eventuale interazione aspecifica anticorpo immunoprecipitante/rab7 che, qualora ci fosse, ti darebbe dei falsi positivi.
Le lane 1 e 2 corrispondono ai lisati totali, gli input che tu usi nella immunprecipitazione. Ti confermano la presenza di rab7 nel sistema (cellule hela) che tu usi. E' un controllo molto importante, perchè se non hai la proteina qui ovviamente non la tirerai giu' manco con la ip (ti esclude quindi i falsi negativi).
3 piccole annotazioni:
1) forse come controllo nella lane 3 sarebbe piu' corretto mettere un trasfettato con plasmide nudo, cioé senza inserto (senza le parti della proteina vimentina). Non ti cambia nulla da un punto di vista sperimentale, ma personalmente lo vedo piu' corretto da un punto di vista formale.
2)dici che la rab7 ti corre in tutte le lanes alla stessa altezza, ma a me nelle lane 4 e 5 mi paiono un po' piu' basse, non so dirti perchè, forse è solo un effetto della lastra (magari è storta) oppure dipende dalla corsa del gel... boh
3) andrebbe inclusa anche la rivelazione della proteina vimentina (o meglio delle porzioni di essa). Sei sicura che le cellule esprimano quantità equivalenti delle due porzioni? Magari il dominio in lane 4 è meno stabile, viene degradato piu' facilmente e di conseguenza ti precipita meno rab7. Oppure hai immunoprecipitato meno in sample rispetto all'altro... Io lo farei
Ciao SpemannOrganizer, e grazie per la risposta così esaustiva  . .
Ora vorrei chiarire delle cose...
1)il plasmide utilizzato è stato fuso con un "HA" che è un epitopo riconosciuto con alta specificità da anticorpi monoclonali anti-HA;
2)credo che il non trasfettato, il controllo appunto, sia rappresentato da un lisato di cellule Hela (ma non ne sono proprio sicura), se fosse così resterebbe sempre un controllo negativo? cosa si potrebbe dire a riguardo?
3)per quanto riguarda la tua annotazione 2, in realtà la rab7 dovrebbe correre in tutte le lanes alla stessa altezza però, credo ci sia stato un problema nella corsa e per questo motivo le bande delle lanes 4 e 5 sono più basse nella lastra.
Aspetto le vostre risposte  |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 24 gennaio 2011 : 12:23:54 Inserito il - 24 gennaio 2011 : 12:23:54




|
| 2) Rimane sempre un controllo negativo. La mia osservazione deriva dal fatto che generalmente come controllo negativo si usano cellule trasfettate con un plasmide vuoto, ma nn credo cambi molto. |
 |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|








