| Autore |
Discussione |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 26 gennaio 2011 : 15:47:27 Inserito il - 26 gennaio 2011 : 15:47:27




|
Ciao a tutti! Sono di nuovo qui per chiedere consiglio su Genetica (in particolare genetica di popolazione) che Ŕ il mio ultimo esame prima della laurea e ho sempre trovato difficoltÓ.
Intanto vi pongo il mio dubbio sull'eterozigositÓ e poi nei prossimi giorni pubblicher˛ una serie di esercizi da me risolti ma non so se son corretti o meno..
veniamo al quesito di oggi:
sul mio libro di genetica dove parla dell'eterozigositÓ fa riferimento ai gruppi sanguigni MN.
Ovviamente dice che nel caso del singolo locus l'eterozigositÓ Ŕ pari a MN ovvero fA/a
ma nel caso in cui si considerino pi¨ locus ci sono vari modi per identificarla.
in particolare reca l'esempio del gene S che Ŕ strettamente associato al gene M/N. Vi Ŕ poi una tabella dove vengono prese in considerazione pi¨ popolazioni e mostra la frequenza delle quattro combinazioni dei due alleli sui due geni (MS,Ms,NS,Ns).
Vi posto la foto della tabella fatta con il cellulare perchŔ non so benecome fare le tabelle qua...


prendiamo in considerazione solo la popolazione degli Ainu (prima riga) dove sono indicati come tipi gametici:
MS 0.024
Ms 0.381
NS 0.247
Ns 0.348
come eterozigositÓ (H)
dai gameti 0.672
dagli alleli 0.438
io so che ho pi¨ di due alleli per quanto riguarda l'eterozigosi devo considerare le possibili combinazioni per calcolarmi la frequenza...
ma in questo caso come devo considerarli visto che mi han dato i tipi gametici??
non capisco come posso calcolare sia H dai gameti che dagli alleli...
Grazie mille per l'aiuto.
|
|
|
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 26 gennaio 2011 : 23:26:15 Inserito il - 26 gennaio 2011 : 23:26:15





|
Per inserire una tabella utilizza il tag code: 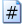 [code ] [ /code] quello che ci scrivi in mezzo rimane allineato. [code ] [ /code] quello che ci scrivi in mezzo rimane allineato.
Dalle foto non si capisce niente, comunque visto che hai scritto i risultati della tabella ti posso rispondere.
Tu hai i gameti quindi puoi fare 2 cose:
1) incrociarli facendo un quadrato di punnet e vedere i genotipi della progenie
ad es. se avessi un gene solo e queste frequenze dei gameti:
A = 0,6 e a= 0,4
i genotipi della progenie sarebbero:
A a
A AA Aa
a Aa aa
e le frequenze le trovi dal prodotto delle frequenze degli alleli quindi:
A a
0,6 0,4
A 0,6 0,36 0,24
a 0,4 0,24 0,16
quindi: AA=0,36 Aa=0,48 aa=0,16
Questo solo per renderti chiari i calcoli, qua ti metto l'immagine di una tabella fatta allo stesso modo con i tuoi dati:
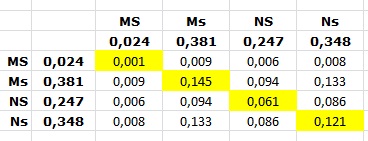
Cosý hai la frequenza di tutti i genotipi, quelli evidenziati in giallo sono omozigoti e non ci interessano.
Ora se sommi le frequenze di tutti gli eterozigoti:
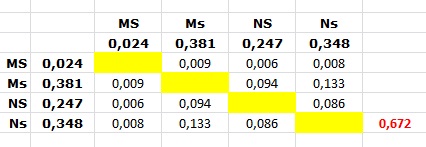
ottieni il valore di "eterozigositÓ dai gameti" 0,672
2) dai gameti puoi anche calcolarti le frequenze alleliche.
Quindi da queste frequenze dei gameti:
MS 0,024
Ms 0,381
NS 0,247
Ns 0,348
Ottieni queste frequenze alleliche:
M 0,405
N 0,595
S 0,271
s 0,729
Poi applicando H-W ottieni le frequenze degli eterozigoti:
Mn = 2pq = 0,482
Ss = 2pq = 0,395
Le sommi e dividi per 2, perchŔ stai considerando 2 geni, e ottieni "l'eterozigositÓ dagli alleli":
H = (0,482 + 0,395)/2 = 0,438
Il concetto Ŕ questo!
 |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 27 gennaio 2011 : 00:09:00 Inserito il - 27 gennaio 2011 : 00:09:00




|
Grazie mille sei stata molto esauriente... finalmente hocapito! grazie 
Se devo esser sincera avevo proprio provato a risolvere il quesito tramite quadrato di punnet ma poi sbagliavo l'approccio aritmetico.. 
per quanto riguarda gli altri esercizi di genetica di popolazione li posso mettere qua senza creare altre discussioni??
magari poi se io non posso mi puoi modificare il titolo in modo tale da mantenere il forum pi├╣ in ordine 
inizio :)
1.Quali sono le forze evolutive che prevengono la speciazione?
MIGRAZIONE, MUTAZIONE, RICOMBINAZIONE
2.Le mutazioni causate da elementi trasponibili sono stabili o instabili?
INSTABILI
3. CATCATCATTCCGGA............
Trasposone ha sequenze invertite di 9 bp e produce duplicazioni del sito target di 6 bp.
Traccia linea sotto ripetizioni invertite e disegna rettangoli attorno al sito target di 6bp (in grassetto).
Completa i 20 nucleotidi mancanti dove non ├Ę possibile.
CATCATCATTCCGGAAGGCCTXXXXXCATCATCAT
4.In una popolazione in equilibrio H-W la frequenza di eterozigoti ├Ę di 6 volte quella degli
omozigoti recessivi. Qual ├Ę la frequenza dell'allele recessivo?
2pq= 6q^2
p+q=1
p=1-q
2(1-q)*q = 6q^2
2q-2q^2 = 6q^2
2q=8q^2
8q^2-2q=0
q=0
q=2/8=1/4
p=3/4
5.I seguenti genotipi per locus RFLP autosomico sono ritrovati in una popolazione:
100 individui totali.
GENE I
OMOZIGOTE DOMINANTE 3.7 kb 0.16
OMOZIGOTE RECESSIVO 2.9 kb 0.36
ETEROZIGOTE 2.9/2.7 kb 0.48
GENE II
OMOZIGOTE D 4.1 kb 0.32
OMOZIGOTE R 3.4 kb 0.44
ETEROZIGOTE 4.1/3.4 kb 0.24
a.frequenze alleliche a ogni locus?
b.frequenze genotipiche se la popolazione ├Ę in equilibrio H-W?
c.quali sono le ragioni per cui un locus pu├▓ essere in H-W e un altro locus indipendente dalla stessa
popolazione pu├▓ non esserlo?
a. GENE I
f allelica A : 0.16 + (1/2)(0.48) = 0.4 p = 40%
f allelica a : 0.36 + (1/2)(0.48) = 0.6 q = 60%
f allelica Aa : 2pq = 2*0.4*0.6 = 0.48 = 48%
GENE II
f allelica A: 0.32 +(1/2)(0.24) = 0.44 = 44%
f allelica a : 0.44 + (1/2)(0.24)= 0.56 = 56 %
f allelica Aa : 2pq = 2* 0.44 * 0.56 = 0.49 = 49 %
GENE II
4.1 kb 0.32
3.4 kb 0.44
4.1/3.4 kb 0.24
b. GENE I
A/A = p^2 = o.4^2 = 0.16
a/a = q^2 = 0.6^2 = 0.36
A/a = 2pq = 2*0.4*0.6 = 0.48
GENE II
A/A = p^2 = o.44^2 = 0.20
a/a = q^2 = 0.56^2 = 0.32
A/a = 2pq = 2*0.44*0.56 = 0.49
c.LINKAGE DISEQUILIBRIUM.
L'equilibrio ├Ę la conseguenza diretta della segregazione degli alleli alla meiosi nell'eterozigosi.
6.Il genoma umano ├Ę lungo 8 miliardi di basi e ogni reazione di sequenziamento pu├▓ determinare
600 basi alla volta. Come mai abbiamo bisogno di 60 milioni di reazioni per coprire l'intero
genoma? Tale sequenza rappresenta veramente il genoma intero della specie? Perch├Ę? Quali sono le
finalità del sequenziamento del genoma?
Perch├Ę per quanto riguarda i progetti di genomica vengono eseguite 10 letture di sequenza indipendenti per tutte le copie di basi del genoma. Cos├Č facendo si ottiene una sequenza corretta poich├ę gli errori casuali non determinano una sua errata ricostruzione. La sequenza corrisponde al genoma intero perch├Ę con questa tecnica si ottiene una sequenza consenso, mettendo insieme le varie letture (assemblaggio delle sequenze) che rappresentano in modo veritiero la sequenza per ogni molecola di DNA del genoma in studio.
Le finalità nel produrre una sequenza consenso sono: far si che sia una rappresentazione reale del genoma in modo da tenere tale sequenza da riferimento per la specie anche se, le differenze tra i vari individui fa si che nessun genoma sia completamente uguale anche in una specie, però si usa come riferimento standard e per quanto riguarda l'individuazione dei geni malattia.
7.Popolazione naturale di piante ad autoimpollinazione viene incrociata. La varianza totale di semi per pianta ├Ę 1.6. 20 piante sono autofecondate per 10 generazioni. Varianza media dopo 10
generazioni ├Ę simile ed ├Ę in media 5.8. stimare H del numero di semi in questa popolazione.
Se^2 = 1.6
Se^2 + Sg^2 = 5.8 Sg^2= 5.8-1.6 = 4.2
H^2 = 4.2/5.8= 0,72
8.Peso corporeo medio dei tacchini ├Ę 5kg. Peso medio aumentato selezionando tacchini con peso medio di 6.2 kg. Progenie ha peso medio di 5.2kg. Determinare h^2.
DIFFERENZIALE DI SELEZIONE= 6.2-5= 1.2 kh = S
RISPOSTA DI SELEZIONE = 5.2-5 =0.2 kg = R
h^2 o EREDITABILITA' IN SENSO STRETTO = 0.2/1.2 = 0.166 =16.6%
9.Se una singola pianta ├Ę eterozigote per 6 loci poligenici additivi che influenzano l'altezza e se
viene autofecondato:
a. quante sono le classi di altezza nella progenie?
b. quale frequenza della classe con le piante pi├╣ alte?
a.6^2=36
b.Frequenza pi├╣ bassa 1/36 ma pi├╣ alta non so come trovare forse 35/36?
10. 7 loci hanno effetto additivo. Determinare il numero di classi fenotipiche e la frequenza fenotipica pi├╣ alta.
7^2 = 49
f fenotipica pi├╣ bassa 1/49 pi├╣ alta forse 48/49?
11.Un girasole ├Ę pi├╣ grande e pi├╣ giallo degli altri nello stesso campo. ├ł della stessa specie?
Si per├▓ ha un corredo genetico diverso essendo dello stesso campo subiscono gli stessi effetti
ambientali.
12.Perch├Ę con l'uso del fago ├Ę pi├╣ facile sequenziare vicino a un promotore?
Ho cercato anche su internet di capire perch├Ę ma non ho capito.
13.La varianza tra generazione P e F1 ├Ę 10.67. La varianza totale ├Ę 42.69. calcola H^2.
Se^2= 10.67
Se^2+Sg^2=42.69 Sg^2=49.69-10.67=32.02
H^2=32.02/42.69
13.In una popolazione si calcola che la frequenza di fitness di un genotipo recessivo sia 0.90 e che il tasso di mutazione all'allele recessivo sia 5*10^-5. posto che la popolazione sia in equilibrio H-W,
quali sono le frequenze alleliche?
Q^ = radice quadrata (mu/s)
mu= 5*10^-5
s= 1-W = 1-0,90 ) 0.10
quindi q^= (5*10^-5)/0.10 = 2.2 *10^-2
p^ = 1-0,022= 0.978
14.Forze che fan cambiare la frequenza allelica in una popolazione:
selezione naturale, mutazione, migrazione, accoppiamento non casuale e deriva genetica.
15.in una popolazione vi sono due alleli del locus A (A1 e A2). Test rivelano che ci sono 384 con A1/A1, 210 con A1/A2 e 260 con A2/A2. Determina le frequenze dei due alleli.
Frequenze genotipiche
A1/A1 = 384/854
A1/A2= 210/854
A2/A2= 260/854
f A1= [(384*2) + (1/2)210]/ (854*2) = 0.51
fA2 = p+q = 1 q=1-p = 1-0.51 = 0.49
16.In una popolazione individui con 4% corpo nero (b) e 96% corpo bianco (B) in equilibrio H-W-
determinare frequenze alleliche Be b e frequenze genotipiche B/B e B/b.
Q^2= 0.4 q=0.64
p=1-0.64=0.36
quindi:
p^2= 0.36^2 = 0.13 (B/B)
q^2= 0.4 (b/b)
2pq = 2* 0.36*0.64 = 0.46
frequenza allelica B : 0.36+(1/2)0.46 = 0.60
frequenza allelica b: 0.4+ (1/2)0.46= 0.63
17.La fitness di 3 genotipi ├Ę
W (A/A) =0.9
W(A/a) = 1
W(a/a) = 0.7
a.se la popolazione parte da F allelica di p= 0.5 qual ├Ę il p' dopo una generazione?
b.qual ├Ę la frequenza allelica all'equilibrio?
pensavo tipo:
p=0.5 quindi q=1-p= 0.5
AA=p^2=0.5^2=0.25
aa=q^2=0.5^2=0.25
A/a=2pq=2*0.25=0.50
devo ricalcolare le frequenze in base alla fitness
AA=p^2*WAA= 0.25*0.9= 0.225
aa=q^2*Waa= 0.25*0.7 = 0.175
Aa= 2pq*WAa= 0.50*1 = 0.50
ora devo correggere le frequenze sulla pase della fitness media data dalla somma delle frequenze calcolate sopra ovvero 0.9
quindi :
AA=p^2*(WAA/Wmedio)=0.25
aa=q^2*(Waa/Wmedio)= 0.20
Aa= 2pq*(WAa/Wmedio)= 0.55
ora posso trovare p'
p'= AA + 1/2 Aa = [p^2(Waa/Wmedio]+ 1/2[2pq(WAa/Wmedio)]= 0.25 + 0.275= 0.525
18.H^2 per alcuni caratteri della vacca sono:
-% proteine latte 0.54
-efficienza nutrizione 0.34
-produzione latte 0.30
-lunghezza intervallo di gestazione 0.01
quale risponde meglio a una selezione artificiale e quale meno?
Proteine del latte risponde meglio mentre lunghezza intervallo gestazione meno poich├ę pi├╣ si avvicina a 1 pi├╣ grande ├Ę la porzione di varianza fenotipica totale da varianza genetica.
19.In una popolazione di scarafaggi la varianza totale del peso del corpo ├Ę 130. si ├Ę stimato che la varianza di dominanza sia pari a 35 e la varianza genetica di dominanza sia pari a 45. Calcolare
l'ereditabilità in senso stretto.
20. simile a es. 5
GENE I
OD 3.3 kb 0.49
OR 4.1 kb 0.09
E 3.3/4.1 kb 0.42
GENE I
p^2= 0.49^2= 0.24
q^2= 0.09^2= 0.0081
2pq= 2*0.24*0.0081= 0.00389
Fallelica
fA/A= 0.49+1/2(0.42)= 0.7 p
fa/a= 0.09+1/2(0.42)= 0.3 q
Fgenotipica
AA= p^2= 0.49
aa= q^2= 0.09
GENE II
3.3 kb 0.04
3.9 kb 0.64
3.3/3.9 kb 0.32
GENE II
p^2= 0.04^2= 0.0016
q^2=0.64^2= 0.40
2pq= 2*0.0016*0.40= 0.00128
Fallelica
FA/A= 0.04+1/2(0.32)= 0.20
Fa/a= 0.64+1/2(0.32)= 0.80
Fgenotipica
AA= p^2= 0.04
aa=q^2=0.64
la popolazione ├Ę in equilibrio H-W? SI perch├Ę la somma di q+p da 1
Cosa succede se individui della popolazione 2 vanno in popolazione 1?
AA e Aa diminuiscono mentre aa aumenta |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 27 gennaio 2011 : 20:41:28 Inserito il - 27 gennaio 2011 : 20:41:28





|
Nessun problema a mettere qua altri esercizi, per˛ il fatto Ŕ che 20 sono veramente tanti!!!
Diventa difficile risponderti!
Va bene utilizzare il forum per chiarire alcuni dubbi, ma Ŕ difficile che qualcuno si metta a risponderti a 20 esercizi, magari seleziona un po' e scrivi quali sono i tuoi dubbi pi¨ grossi. |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 27 gennaio 2011 : 23:22:58 Inserito il - 27 gennaio 2011 : 23:22:58




|
ok scusa ├Ę che quegli esercizi verranno messi all'esame per quello speravo che mi venisse detto se ero sulla buona strada o meno anche perch├Ę non ho modo di correggerli sola...
Comunque..
1. CATCATCATTCCGGA............
Trasposone ha sequenze invertite di 9 bp e produce duplicazioni del sito target di 6 bp.
Traccia linea sotto ripetizioni invertite e disegna rettangoli attorno al sito target di 6bp (in grassetto).
Completa i 20 nucleotidi mancanti dove non ├Ę possibile.
CATCATCAT TCCGGA AGGCCTXXXXXCATCATCAT
seq.inv. target nucleotidi mancanti
2.In una popolazione in equilibrio H-W la frequenza di eterozigoti ├Ę di 6 volte quella degli
omozigoti recessivi. Qual ├Ę la frequenza dell'allele recessivo?
2pq= 6q^2
p+q=1
p=1-q
2(1-q)*q = 6q^2
2q-2q^2 = 6q^2
2q=8q^2
8q^2-2q=0
q=0
q=2/8=1/4
p=3/4
3.I seguenti genotipi per locus RFLP autosomico sono ritrovati in una popolazione:
100 individui totali.
GENE I
OMOZIGOTE DOMINANTE 3.7 kb 0.16
OMOZIGOTE RECESSIVO 2.9 kb 0.36
ETEROZIGOTE 2.9/2.7 kb 0.48
GENE II
OMOZIGOTE D 4.1 kb 0.32
OMOZIGOTE R 3.4 kb 0.44
ETEROZIGOTE 4.1/3.4 kb 0.24
a.frequenze alleliche a ogni locus?
b.frequenze genotipiche se la popolazione ├Ę in equilibrio H-W?
c.quali sono le ragioni per cui un locus pu├▓ essere in H-W e un altro locus indipendente dalla stessa
popolazione pu├▓ non esserlo?
a. GENE I
f allelica A : 0.16 + (1/2)(0.48) = 0.4 p = 40%
f allelica a : 0.36 + (1/2)(0.48) = 0.6 q = 60%
f allelica Aa : 2pq = 2*0.4*0.6 = 0.48 = 48%
GENE II
f allelica A: 0.32 +(1/2)(0.24) = 0.44 = 44%
f allelica a : 0.44 + (1/2)(0.24)= 0.56 = 56 %
f allelica Aa : 2pq = 2* 0.44 * 0.56 = 0.49 = 49 %
GENE II
4.1 kb 0.32
3.4 kb 0.44
4.1/3.4 kb 0.24
b. GENE I
A/A = p^2 = o.4^2 = 0.16
a/a = q^2 = 0.6^2 = 0.36
A/a = 2pq = 2*0.4*0.6 = 0.48
GENE II
A/A = p^2 = o.44^2 = 0.20
a/a = q^2 = 0.56^2 = 0.32
A/a = 2pq = 2*0.44*0.56 = 0.49
c.LINKAGE DISEQUILIBRIUM.
L'equilibrio ├Ę la conseguenza diretta della segregazione degli alleli alla meiosi nell'eterozigosi.
4.Se una singola pianta ├Ę eterozigote per 6 loci poligenici additivi che influenzano l'altezza e se
viene autofecondato:
a. quante sono le classi di altezza nella progenie?
b. quale frequenza della classe con le piante pi├╣ alte?
a.6^2=36
b.Frequenza pi├╣ bassa 1/36 ma pi├╣ alta non so come trovare forse 35/36?
5a.Un girasole ├Ę pi├╣ grande e pi├╣ giallo degli altri nello stesso campo. ├ł della stessa specie?
Si per├▓ ha un corredo genetico diverso essendo dello stesso campo subiscono gli stessi effetti
ambientali.
b.Perch├Ę con l'uso del fago ├Ę pi├╣ facile sequenziare vicino a un promotore?
Ho cercato anche su internet di capire perch├Ę ma non ho capito.
6.La fitness di 3 genotipi ├Ę
W (A/A) =0.9
W(A/a) = 1
W(a/a) = 0.7
a.se la popolazione parte da F allelica di p= 0.5 qual ├Ę il p' dopo una generazione?
b.qual ├Ę la frequenza allelica all'equilibrio?
pensavo tipo:
p=0.5 quindi q=1-p= 0.5
AA=p^2=0.5^2=0.25
aa=q^2=0.5^2=0.25
A/a=2pq=2*0.25=0.50
devo ricalcolare le frequenze in base alla fitness
AA=p^2*WAA= 0.25*0.9= 0.225
aa=q^2*Waa= 0.25*0.7 = 0.175
Aa= 2pq*WAa= 0.50*1 = 0.50
ora devo correggere le frequenze sulla pase della fitness media data dalla somma delle frequenze calcolate sopra ovvero 0.9
quindi :
AA=p^2*(WAA/Wmedio)=0.25
aa=q^2*(Waa/Wmedio)= 0.20
Aa= 2pq*(WAa/Wmedio)= 0.55
ora posso trovare p'
p'= AA + 1/2 Aa = [p^2(Waa/Wmedio]+ 1/2[2pq(WAa/Wmedio)]= 0.25 + 0.275= 0.525
7..H^2 per alcuni caratteri della vacca sono:
-% proteine latte 0.54
-efficienza nutrizione 0.34
-produzione latte 0.30
-lunghezza intervallo di gestazione 0.01
quale risponde meglio a una selezione artificiale e quale meno?
Proteine del latte risponde meglio mentre lunghezza intervallo gestazione meno poich├ę pi├╣ si avvicina a 1 pi├╣ grande ├Ę la porzione di varianza fenotipica totale da varianza genetica.
8.In una popolazione di scarafaggi la varianza totale del peso del corpo ├Ę 130. si ├Ę stimato che la varianza di dominanza sia pari a 35 e la varianza genetica di dominanza sia pari a 45. Calcolare
l'ereditabilità in senso stretto.
sp^2= 130
e poi non so come considerare la varianza di dominanza e la varianza genetica di dominanza...
so che dovrei trovare sg^2 e se^2 per poi applicare la formula
H^2= Sg^2/Sp^2 = Sg^2/(Sg^2+Se^2)
Ho eliminato ben 12 esercizi
 |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 01 febbraio 2011 : 07:39:10 Inserito il - 01 febbraio 2011 : 07:39:10




|
tra una settimana e mezza ho l'esame nessuno riesce ad indicarmi se sono sulla via giusta con questi esercizi?
grazie mille  |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 02 febbraio 2011 : 07:51:12 Inserito il - 02 febbraio 2011 : 07:51:12




|
ok prover˛ a vedere...
nel caso del secondo esercizio c'era la domanda anche cosa succede nel caso in cui alcuni individui con il gene II si mescolano con alcuni individui con il gene I.
ho pensato di guardare le frequenze alleliche:
GENE I
f allelica A : 0.16 + (1/2)(0.48) = 0.4 p = 40%
f allelica a : 0.36 + (1/2)(0.48) = 0.6 q = 60%
f allelica Aa : 2pq = 2*0.4*0.6 = 0.48 = 48%
GENE II
f allelica A: 0.32 +(1/2)(0.24) = 0.44 = 44%
f allelica a : 0.44 + (1/2)(0.24)= 0.56 = 56 %
f allelica Aa : 2pq = 2* 0.44 * 0.56 = 0.49 = 49 %
e di rispondere cosý:
nel caso della fallelica A aumenterÓ leggermente
nel caso della fallelica a diminuirÓ leggermente
nel caso della fallelica Aa resterÓ pressochŔ invariato con una leggera tendenza ad aumentare. |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 02 febbraio 2011 : 10:14:29 Inserito il - 02 febbraio 2011 : 10:14:29




|
vediamo se ho capito l'esercizio 1
1. CATCATCATTCCGGA............
Trasposone ha sequenze invertite di 9 bp e produce duplicazioni del sito target di 6 bp.
Traccia linea sotto ripetizioni invertite e disegna rettangoli attorno al sito target di 6bp (in grassetto).
Completa i 20 nucleotidi mancanti dove non ├Ę possibile.
CATCATCAT TCCGGA XXXXXAGGCCTCATCATCAT
seq.inv. target nucleotidi mancanti |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 02 febbraio 2011 : 23:46:20 Inserito il - 02 febbraio 2011 : 23:46:20





|
Citazione:
Messaggio inserito da lunatuning
vediamo se ho capito l'esercizio 1
1. CATCATCATTCCGGA............
Trasposone ha sequenze invertite di 9 bp e produce duplicazioni del sito target di 6 bp.
Traccia linea sotto ripetizioni invertite e disegna rettangoli attorno al sito target di 6bp (in grassetto).
Completa i 20 nucleotidi mancanti dove non ├Ę possibile.
CATCATCAT TCCGGA XXXXXAGGCCTCATCATCAT
seq.inv. target nucleotidi mancanti
Ma l'hai riscritto uguale a prima!
Hai letto i file che ti linkato?
Cito dal secondo link:
Citazione:
Dal momento che le estremitÓ di una sequenza di inserzione sono invertite, se una estremitÓ della sequenza di inversione Ŕ 5'-ACCGTAG, lĺaltra estremitÓ sarÓ quella complementare invertita: CTACGGT-3'.
vedi anche fig. 21.1
Tu dovresti avere "sequenze invertite di 9 bp", non le hai scritte invertite!
Secondo: le sequenze invertite sono nel trasposone, quindi "interne", mentre il sito target Ŕ dove si inserisce il trasposone, quindi ovviamente "esterno", tu hai scritto il tutto esattamente al contrario.
Le ripetizioni nel sito target sono dirette, vedi fig. 21.2
il che vuol dire che se hai un sito target ad es. AAGTCG quando si inserisce il trasposone hai:
AAGTCG-TRASPOSONE-AAGTCG |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 03 febbraio 2011 : 00:03:46 Inserito il - 03 febbraio 2011 : 00:03:46





|
Per l'esercizio 3:
Citazione:
a. GENE I
f allelica A : 0.16 + (1/2)(0.48) = 0.4 p = 40%
f allelica a : 0.36 + (1/2)(0.48) = 0.6 q = 60%
f allelica Aa : 2pq = 2*0.4*0.6 = 0.48 = 48%
gli alleli sono 2 "A" e "a", "Aa" Ŕ un genotipo (come AA e aa)!
Quello che hai calcolato come 2pq Ŕ la frequenza del genotipo Aa.
Per il gene II ovviamente vale lo stesso discorso!
La risposta b. Ŕ giusta (anche se per un problema di appossimazione nel gene II il totale non fa 100% ma 101%)
Per la c. il linkage disequilibrium va bene come spiegazione, la frase che hai scritto dopo non l'ho capita.
Poi nell'ultima tua risposta dove dici: "nel caso del secondo esercizio c'era la domanda anche cosa succede nel caso in cui alcuni individui con il gene II si mescolano con alcuni individui con il gene I."
penso ti riferissi sempre a questo esercizio 3.
Comunque non ha senso la domanda, sicura fosse cosý?
Sono 2 geni diversi e ogni individuo avrÓ una determinata coppia di alleli per ciascun gene, ma avrÓ entrambi i geni, non possono esistere individui con solo il gene I e individui con solo il gene II.
Per il 4:
Citazione:
4.Se una singola pianta Ŕ eterozigote per 6 loci poligenici additivi che influenzano l'altezza e se
viene autofecondato:
a. quante sono le classi di altezza nella progenie?
Citazione:
a.6^2=36
6^2 da dove Ŕ venuto fuori?
Se hai "n" loci poligenici, ciascuno con 2 alleli avrai.
n░ ALLELI = 2n
n░ genotipi= 3^n
n░ fenotipi= 2n+1
quindi?
Potresti anche contarli a mano, ma Ŕ lunga, ti faccio l'esempio con 2 geni.
n░ geni= 2 (A e B)
n░ ALLELI = 2n = 4 (A, a, B e b)
n░ genotipi= 3^n = 3^2 = 9 (AABB, AABb, AAbb, AaBB, AaBb, Aabb, aaBB, aaBb, aabb)
n░ fenotipi= 2n+1 = 5
4 alleli dominati: AABB
3 alleli dominati: AABb o AaBB
2 alleli dominati: AAbb o aaBB o AaBb
1 alleli dominati: Aabb o aaBb
0 alleli dominati: aabb Citazione:
b.Frequenza pi¨ bassa 1/36 ma pi¨ alta non so come trovare forse 35/36?
Come hai calcolato 1/36?
Comunque come vedi anche dallo schema sopra le piante pi¨ alte e quelle pi¨ basse sono quelle con i genotipi estremi:
0 alleli dominati: piante pi¨ basse
tutti alleli dominati: piante pi¨ alte
e la frequenza Ŕ identica!
La frequenza del genotipo estremo la trovi come: (1/2)^2n
Se ti chiedesse anche le frequenze dei genotipi intermedi il denominatore lo calcoli come sopra (ovviamente sarÓ lo stesso) e per il numeratore usi il triangolo di Tartaglia:

L'immagine si ferma a 10, tu dovresti arrivare a 12 perchÚ hai 12 alleli. Di certo non ti faranno una domanda del genere all'esame, ma Ŕ giusto saperlo! |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 03 febbraio 2011 : 23:50:37 Inserito il - 03 febbraio 2011 : 23:50:37




|
scusami sto cercando di capire l'esercizio 4 anche in base ai miei appunti...
quindi
n░ alleli = 64
n░ genotipi= 729
n░ fenotipi= 5
non essendo intermedi ma genotipi estremi ovvero o piante alte o piante basse uso la formula che mi hai indicato:
(1/2)^2n = 0.00024 |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 04 febbraio 2011 : 00:00:47 Inserito il - 04 febbraio 2011 : 00:00:47




|
CATCAT CATTCCGGA XXXXX CATTCCGGA TACTAC
TARGET SEQ.INVERT nucleotidi mancanti
ho capito o son ancora fuori strada?
Ŕ tardi e oggi avevo un esame se Ŕ sbagliato cerca di avere pazienza non lo faccio con cattiveria a non capire...
i link li ho letti ma sul mio libro non Ŕ spiegato proprio cosý... |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 07 febbraio 2011 : 16:25:53 Inserito il - 07 febbraio 2011 : 16:25:53




|
Citazione:
Messaggio inserito da lunatuning
7..H^2 per alcuni caratteri della vacca sono:
-% proteine latte 0.54
-efficienza nutrizione 0.34
-produzione latte 0.30
-lunghezza intervallo di gestazione 0.01
quale risponde meglio a una selezione artificiale e quale meno?
Proteine del latte risponde meglio mentre lunghezza intervallo gestazione meno poich├ę pi├╣ si avvicina a 1 pi├╣ grande ├Ę la porzione di varianza fenotipica totale da varianza genetica.
ho detto una cavolata.....
pi¨ Ŕ elevata h^2 maggiore correlazione avr˛ tra genitori e figli
quindi per la selezione artificiale il carattere che risponderÓ meglio sarÓ la % delle proteine del latte mentre la lunghezza dell'intervallo di gestazione meno
|
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 07 febbraio 2011 : 17:53:13 Inserito il - 07 febbraio 2011 : 17:53:13





|
Citazione:
Messaggio inserito da lunatuning
CATCAT CATTCCGGA XXXXX CATTCCGGA TACTAC
TARGET SEQ.INVERT nucleotidi mancanti
ho capito o son ancora fuori strada?
TARGET:
cito dal secondo link:
Citazione:
Unĺaltra caratteristica di una sequenza di inserzione, in comune con molti trasposoni pi¨ complessi, si trova appena fuori dal trasposone stesso. Si tratta di un paio di corte sequenze ripetute dirette, nella regione di DNA immediatamente
circostante il trasposone. Queste ripetizioni non si osservano se non
dopo che il trasposone si Ŕ inserito; risultano dal processo stesso di inserzione e suggeriscono che la trasposasi tagli il DNA in maniera sfalsata piuttosto che tramite due tagli uno di fronte allĺaltro. La Figura 21.2 mostra come i tagli sfalsati sui due filamenti di DNA nel sito dellĺinserzione conducano automaticamente
alla ripetizione diretta.
figura 21.2
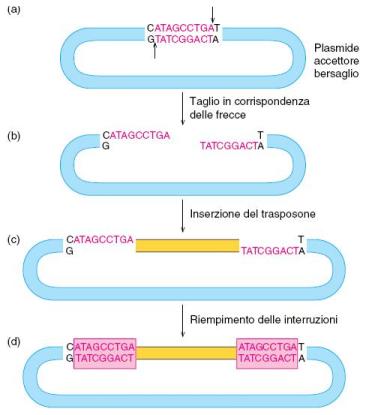
La ripetizione Ŕ diretta, se il tuo target Ŕ CATCAT
avrai CATCAT...TRASPOSONE...CATCAT
non CATCAT...TRASPOSONE...TACTAC
SEQUENZE INVERTITE:
sono appunto "invertite" come ti dice il nome, tu le hai scritte identiche:
Citazione:
CATCAT CATTCCGGA XXXXX CATTCCGGA TACTAC
TARGET SEQ.INVERT nucleotidi mancanti
dal link che ti avevo anche giÓ citato:
Citazione:
Dal momento che le estremitÓ di una sequenza di inserzione sono invertite, se una estremitÓ della sequenza di inversione Ŕ 5'-ACCGTAG, lĺaltra estremitÓ sarÓ quella complementare invertita: CTACGGT-3'.
se ACCGTAG diventa CTACGGT
la tua che Ŕ: CATTCCGGA
cosa diventa?
P.S. tu che libro hai? |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 07 febbraio 2011 : 18:08:40 Inserito il - 07 febbraio 2011 : 18:08:40





|
Citazione:
Messaggio inserito da lunatuning
scusami sto cercando di capire l'esercizio 4 anche in base ai miei appunti...
quindi
n░ alleli = 64
n░ genotipi= 729
n░ fenotipi= 5
non essendo intermedi ma genotipi estremi ovvero o piante alte o piante basse uso la formula che mi hai indicato:
(1/2)^2n = 0.00024
Il numero di alleli Ŕ 2n non 2^n!
2 x 6 = 12
(potresti anche contarli, per 6 geni: A, B, C, D, E, F
hai questi alleli: A, a, B, b, C, c, D, d, E, e, F, f)
Il numero di genotipi l'hai scritto giusto.
Il numero di "fenotipi" Ŕ 2n+1
quindi 2 x 6 + 1 = 13
come vedi non Ŕ altro che il numero di alleli pi¨ 1 (come era comprensibile anche dalla tabella che ti avevo scritto per 2 geni)
La frequenza dei genotipi estremi Ŕ giusta 0.00024 (o 1/4096).
Sý quelli che ti chiede sono quelli estremi, io ti ho aggiunto anche la spiegazione per gli altri, anche sae non c'era come domanda. |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 07 febbraio 2011 : 18:14:36 Inserito il - 07 febbraio 2011 : 18:14:36




|
giuro non capisco come fan a diventare cos├Č
se ACCGTAG diventa CTACGGT
io pensavo che per invertite volesse dire che se ho ACCGTAG diventa GATGCCA
infatti in base ai link avevo pensato a una possibile risoluzione dell'esercizio tipo:
CATCAT|CATTCCGGA
GTAGTA GTAAGGCCT|
CATCAT CATTCCGGA
GTCGTC GTAAGGCCT
CATCAT CATTCCGGA
GTCGTC GTAAGGCCT
CATCAT CATTCCGGA CATTCCGGA
GTCGTC GTAAGGCCT CATTCCGGA
CATCAT CATTCCGGA XXXXX CATTCCGGA
GTCGTC GTAAGGCCT XXXXX CATTCCGGA
|
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 07 febbraio 2011 : 18:27:25 Inserito il - 07 febbraio 2011 : 18:27:25




|
gli altri 4 esercizi son giusti?
a breve avr├▓ l'esame e sto studiando veramente tanto.. solo che non mi trovo bene con questo libro di genetica perch├Ę spiega un po' veramente male |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 07 febbraio 2011 : 18:31:07 Inserito il - 07 febbraio 2011 : 18:31:07





|
Citazione:
Messaggio inserito da lunatuning
giuro non capisco come fan a diventare cos├Č
se ACCGTAG diventa CTACGGT
Sono "invertite" e "complementari":
ACCGTAG
lo inverti, ovvero lo leggi al contrario: GATGCCA
poi fai il complementare:
GATGCCA sequenza
CTACGGT complementare
quindi ACCGTAG Ŕ diventato CTACGGT.
Ora prendi la tua: CATTCCGGA
e fai esattamente la stessa cosa.
Visto che hai detto che sul tuo libro Ŕ spiegato diversamente, mi potresti dire qual Ŕ il libro, almeno se ce l'ho posso guardare come Ŕ spiegato? |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 07 febbraio 2011 : 18:38:34 Inserito il - 07 febbraio 2011 : 18:38:34




|
il libro ├Ę "genetica, principi di analisi formale" della zanichelli
oddio ├Ę vero... scusami che figuracce... ├Ę che sono veramente fusa!!!
CATCAT|CATTCCGGA
GTAGTA GTAAGGCCT|
dovrei avere:
CATCAT | CATTCCGGA XXXXX TCCGGTTAC CATCAT |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 07 febbraio 2011 : 18:50:41 Inserito il - 07 febbraio 2011 : 18:50:41





|
Non ho fatto a tempo a vedere tutti gli esercizi, ci vuole un po' e non ho moltissimo tempo.
Ho guardato il 6:
Citazione:
6.La fitness di 3 genotipi Ŕ
W (A/A) =0.9
W(A/a) = 1
W(a/a) = 0.7
a.se la popolazione parte da F allelica di p= 0.5 qual Ŕ il p' dopo una generazione?
b.qual Ŕ la frequenza allelica all'equilibrio?
pensavo tipo:
p=0.5 quindi q=1-p= 0.5
AA=p^2=0.5^2=0.25
aa=q^2=0.5^2=0.25
A/a=2pq=2*0.25=0.50
devo ricalcolare le frequenze in base alla fitness
AA=p^2*WAA= 0.25*0.9= 0.225
aa=q^2*Waa= 0.25*0.7 = 0.175
Aa= 2pq*WAa= 0.50*1 = 0.50
ora devo correggere le frequenze sulla pase della fitness media data dalla somma delle frequenze calcolate sopra ovvero 0.9
quindi :
AA=p^2*(WAA/Wmedio)=0.25
aa=q^2*(Waa/Wmedio)= 0.20
Aa= 2pq*(WAa/Wmedio)= 0.55
ora posso trovare p'
p'= AA + 1/2 Aa = [p^2(Waa/Wmedio]+ 1/2[2pq(WAa/Wmedio)]= 0.25 + 0.275= 0.525
giusto, a parte che alla fine hai sbagliato il secondo valore, hai preso il valore di Aa per la sua fitness anzichŔ questo valore diviso per la fitness media. In sostanza nella formula hai scritto "0.275" al posto di "0.55".
Inoltre, anche se non Ŕ gravissimo, l'approssimazione che hai utilizzato non Ŕ proprio correttissima.
Tu hai scritto:
AA=p^2*(WAA/Wmedio)=0.25
aa=q^2*(Waa/Wmedio)= 0.20
Aa= 2pq*(WAa/Wmedio)= 0.55
per˛ sarebbe:
AA=p^2*(WAA/Wmedio)=0.25
aa=q^2*(Waa/Wmedio)= 0.19444444...
Aa= 2pq*(WAa/Wmedio)= 0.5555555...
quindi meglio scriverli come 0.195 e 0.555
alla fine ti viene p'= 0.68 |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 07 febbraio 2011 : 19:03:46 Inserito il - 07 febbraio 2011 : 19:03:46




|
quello del trasposone l'ho capito comunque??
Grazie mille intanto! |
 |
|
|
GFPina
Moderatore
    

CittÓ: Milano

8408 Messaggi |
 Inserito il - 07 febbraio 2011 : 19:20:36 Inserito il - 07 febbraio 2011 : 19:20:36





|
Ok ho controllato sul tuo libro per quanto riguarda i trasposoni.
In effetti non ti spiega bene come sono le sequenze invertite e l'immagine non Ŕ molto d'aiuto.
Non so se abbiamo la stessa edizione, la mia Ŕ la quinta, la tua magari Ŕ pi¨ nuova e le pagine non corrispondono.
Comunque la figura che ti fa vedere la serquenze IS (che per me Ŕ la 20.14) Ŕ questa:
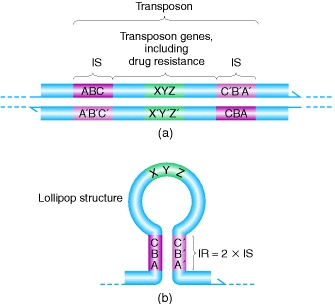
In effetti con ABC non Ŕ molto chiaro, ma il segno ' starebbe per compŔlementare, quindi A'B'C' Ŕ il complementare di ABC e C'B'A' Ŕ il complementare invertito.
Per quanto riguarda la sequenza target, hai un'immagine (per me Ŕ la 20.27) che Ŕ analoga a quella del link che ti ho indicato io, l'immagine Ŕ questa:
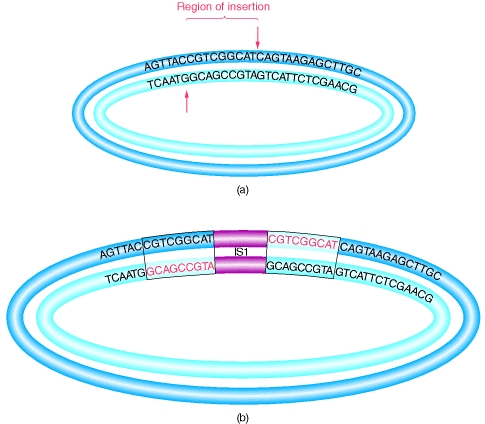
e nel paragrafo "Le conseguanze molecolari della trasposizione" Ŕ spiegato.
Comunque sia:
Tu avevi questa sequenza: CATCATCATTCCGGA
l'hai divisa giusta:
CATCAT | CATTCCGGA
Ma la sequenza invertita l'hai scritta ancora sbagliata:
CATCAT | CATTCCGGA XXXXX TCCGGTTAC CATCAT sarebbe:
CATCAT | CATTCCGGA XXXXX TCCGGAATG CATCAT
|
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 07 febbraio 2011 : 19:39:45 Inserito il - 07 febbraio 2011 : 19:39:45




|
si le ultime le ho sbagliate nella fretta di rispondere... effettivamente non abbiamo la stessa edizione perch├Ę io on ho quelle figure...
nel caso la sequenza fosse stata questa:
CATCAT | CATTCCGGA AGTCAGGA
sarebbe stato come l'esercizio visto che c'├Ę ancora un pezzo di DNA?
CATCAT | CATTCCGGA XXXXX AGTCAGGA TCCGGAATG CATCAT ?
oppure il resto del DNA lo lascio legato alla seguenza come in origine? ovvero:
CATCAT | CATTCCGGA AGTCAGGA XXXXX TCCGGAATG CATCAT |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 09 febbraio 2011 : 18:48:31 Inserito il - 09 febbraio 2011 : 18:48:31




|
posso fare un'ulteriore domanda per chiarirmi le idee??
allora sto studiando il capitolo di genetica medica e mi sono imbattuta nel mappaggio genico.
vediamo se ho capito bene:
il mappaggio genico mi serve per individuare i geni malattia. Si effettua tramite analisi di linkage che richiedono dei marcatori particolari in posizione nota che presentano una certa associazione (consegregazione) con il mio gene malattia, in particolare per quanto riguarda gli alleli.
Ci sono vari tipi di marcatori genetici tra cui:
-RFLP: evidenziati tramite PRC e digestione enzimatica oppure tramite digestione enzimatica e successivo Southern Blot;
-VNTR e Microsatellite: i primi sono identificabili mediante Southern Blot e secondi tramite PCR
-SNP: identificabili per PCR.
Per identificare un gene malattia si pu├▓ procedere anche tramite CLONAGGIO che si distingue in:
-POSIZIONALE: clono un gene sulla base della sua posizione, eseguo analisi di linkage (in pratica ├Ę il mappaggio genico)
-FUNZIONALE: in base alla funzione risalgo al gene (uso modelli animali)
Per quanto riguarda il clonaggio posizionale posso effettuare :
-HIERARCHICAL SEQUENCING: ovvero visualizzo il bandeggio tramite colorazione del cromosoma, una banda di 5 milioni di copie viene rimossa e isolata, il DNA viene tagliato grazie a un'enzima di restrizione e poi viene clonato tramite vettori BAC. Le sequence tagger sites (STS) sono localizzati sui frammenti (quelli comuni sono sedi di sovrapposizione) ed infine i frammenti BAC vengono tagliati in piccoli segmenti e sequenziati da un STS a un STS.
-SHOTGUN SEQUENCING: ha come obiettivo la creazione di enormi dati di sequenza contigua di DNA costruendo quindi una banca di sequenza genomica (ovvero libreria genomica???)
In ogni caso sia con la prima che con la seconda tecnica ottengo pi├╣ geni nella regione considerata e quindi devo identificare il vero gene malattia in vari modi (se conosco il prodotto mi comporter├▓ in un modo se non lo conosco mi comporter├▓ in un altro e ancora se conosco i sintomi).
Per identificare i geni posso anche utilizzare il metodo della cattura degli Esoni...
ma ora il mio dubbio ├Ę ... queste tecniche sono tutte collegate tra loro oppure per l'identificazione o uso il clonaggio o la cattura degli esoni?
queste tecniche comunque alla fine mi servono per arrivare al mapping cromosomico giusto?
grazie mille |
 |
|
|
lunatuning
Nuovo Arrivato

76 Messaggi |
 Inserito il - 14 febbraio 2011 : 14:20:31 Inserito il - 14 febbraio 2011 : 14:20:31




|
gioved├Č ho l'esame help  |
 |
|
| |
Discussione |
|













