Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
karolka
Nuovo Arrivato

2 Messaggi |
 Inserito il - 13 aprile 2012 : 13:50:55 Inserito il - 13 aprile 2012 : 13:50:55




|
Ciao a tutti!
Sono nuova qui sul forum quindi spero di essere nella sezione giusta.
Per il corso di genetica molecolare della mia università mi è stato assegnato un compito riguardo alla costruzione di una mappa di restrizione.
Credo di aver capito bene il concetto (perlomeno leggendo sui libri!), tuttavia ora mi trovo in difficoltà e spero possiate aiutarmi.
Ho i risultati prodotti in seguito all'elettroforesi su gel e cercherò di allegarli assieme al post.
Per quanto riguarda la prima colonna (M) il mio laboratorio usa una scaletta di referenza (hyperladder) per calcolare bP dei frammenti prodotti.
Ho calcolato quindi i seguenti risultati per ogni colonna:
u/c (uncut sequence)=2 frammenti di cui uno 10000bP e l'altro 1995bp
H (hindIII)= un frammento di 3981bP
E (ecoRI)= due frammenti di cui uno 3162bP e l'altro 251bP
E+H (double digest)= 3 frammenti di cui il primo è 3162bP, il secondo 398 e il terzo 251.
calcolando i valori usando logaritmi in base 10 i valori dovrebbero quindi essere
u/c= 4 + 3.3= 7.3
H=3.6
E=3.5 + 2.4= 5.9
E&H=3.5 +2.6 + 2.4= 8.5
Ora, da quel che ho capito, le somme dei frammenti di ogni sequenza dovrebbero equivalere a quelli della sequenza intera (uncut). Non riesco a capire come mai, invece, nessuna somma corrisponde al valore iniziale (in questo caso 7.3). Forse sto sbagliando tutto, ma non so proprio più che pesci pigliare, sarei grata se poteste aiutarmi a capire come procedere o dove sbaglio!Grazie
Karolka
ps. mi scuso se la mia terminologia risulta incorretta/insolita ma studio all'estero e può darsi che le mie doti di traduttrice non siano proprio perfette ;)
Immagine:
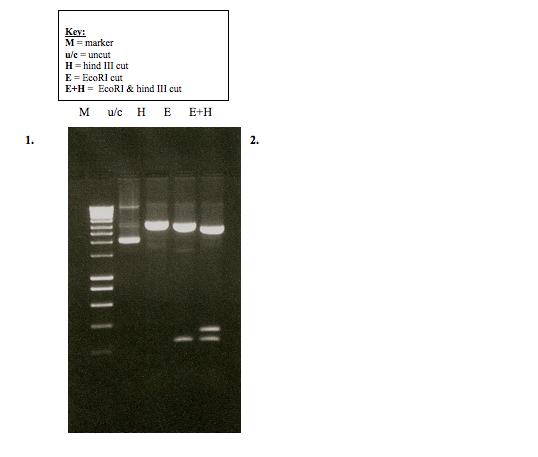
129,64 KB
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 13 aprile 2012 : 22:37:51 Inserito il - 13 aprile 2012 : 22:37:51





|
Di HyperLadder ce ne sono diversi, ma credo che quello che avete utilizzato sia questo: 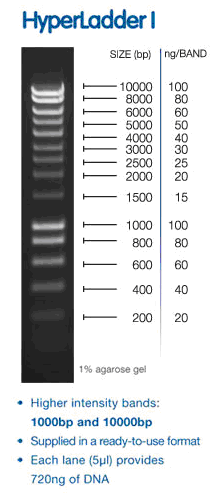
I pesi che hai segnato delle bande non so bene come siano stati calcolati, ma non mi sembrano molto corretti.
Citazione:
Messaggio inserito da karolka
u/c (uncut sequence)=2 frammenti di cui uno 10000bP e l'altro 1995bp
H (hindIII)= un frammento di 3981bP
E (ecoRI)= due frammenti di cui uno 3162bP e l'altro 251bP
E+H (double digest)= 3 frammenti di cui il primo è 3162bP, il secondo 398 e il terzo 251
u/c la banda più intesa potrebbe essere attorno a 1995
ma le due bande più intense di H e di E mi sembrano molto vicine, non credo possano essere H 3981 e E 3162, inoltre nella lane successiva E+H la banda più intensa dici che è ancora 3162, come quella di E, ma non può essere così perché è più bassa. Inoltre così non tornerebbero i conti. Quindi penso che ci sia un errore nelle bande di E.
Poi hai fatto i conti col logaritmo che non capiscono a cosa ti servano, non hanno senso!
Il logaritmo ti serve per calcolare il peso delle bande in bp, che però tu hai già!
La velocità di migrazione su gel è inversamente proporzionale al logaritmo del peso molecolare, quindi facendo un grafico logaritmico con i valori di peso molecolare del marker rispetto alla distanza percorsa (in scala logartimica) ottieni una retta di taratura dalla quale conoscendo la distanza percorsa dai tuoi frammenti incogniti puoi calcolarne il peso molecolare.
Tutto questo però a te non serve perché hai già il peso molecolare, è su quello che devi fare i conti!
Il frammento uncutted non ti dà alcuna indicazione su quale sia il peso totale perchè si tratta di una plasmide circolare che su gel non migra come un frammento lineare, la banda che vedi più intensa è quella della forma suoeravvolta che migra più velocemente, quindi avrai una banda più bassa (in questo caso 1995bp) rispetto a quella dello stesso frammento in forma lineare.
Il peso effettivo del frammento lo vedi se hai un singolo taglio nel plamide che te lo linearizza, nel tuo caso questo avviene con l'enzima Hind III che fa un taglio solo (un solo frammmento). Il peso del plasmide totale quindi sarà 3981bp. Negli altri due casi in cui hai 2 e 3 frammenti la somma dei frammenti di ogni caso deve darti il peso totale del plasmide. Nel caso di E cme ti dicevo c'è un errore nel calcolo del peso della prima banda che dovrebbe essere più grande quindi i conti non tornano, ma nel caso di E+H la somma dei frammenti: è 3162 + 398 + 251= 3821 che è abbastanza vicina a 3981.
Non so però se la differenza è dovuta al fatto che i pesi dei frammmenti non sono corretti, oppure se è solo perchè essendo estrapolati dal gel non sono "esatti".
Spero sia tutto chiaro ora, se hai altri dubbi chiedi pure.
P.S. curiosità, in che paese studi? |
 |
|
|
karolka
Nuovo Arrivato

2 Messaggi |
 Inserito il - 14 aprile 2012 : 12:28:24 Inserito il - 14 aprile 2012 : 12:28:24




|
Grazie mille per la spiegazione! In effetti ora tutto ha senso!!
Il mio prof di genetica è uno di poche parole e non si sbilancia mai nel dare spiegazioni..e io sono ancora alle prime armi con questi tipi di esperimenti!
Per quanto riguarda E, controllando meglio mi son resa conto che il mio valore è sbagliato, ma ora l' ho corretto :)
I conti con il logaritmo li ho dovuti fare su richiesta dell'esercizio, ed ho fatto anche il grafico..non so dare una spiegazione migliore, mi son limitata a seguire le istruzioni sul mio manuale :D
Poi per quanto riguarda il frammento uncut, quasi mi vergogno a non aver pensato che in effetti, essendo circolare, migra diversamente..grazie per avermi illuminato!!!!
Per quanto riguarda invece il peso di E+H penso che la differenza sia dovuta al fatto che questo metodo non è molto accurato...ma è un problema facilmente risolvibile.
Oggi proverò a finire l'esercizio costruendo la mappa e spero di non avere problemi, nel caso ne approfitterò per chiedere di nuovo ;)
In realtà una domandina prima di procedere ce l'avrei: mi risulta che il plasmide puc18 (che è stato usato in questo caso) abbia solo una sequenza di riconoscimento per ecoR1, e quindi dovrebbe produrre un frammento solo, giusto? Come mai in questo caso ce ne sono 2? Mi viene il dubbio che ci sia un errore nelle "etichette" delle colonne..
Grazie ancora per l'aiuto!
Karolka
ps. studio a Manchester, UK :) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 14 aprile 2012 : 18:13:40 Inserito il - 14 aprile 2012 : 18:13:40





|
Citazione:
Messaggio inserito da karolka
In realtà una domandina prima di procedere ce l'avrei: mi risulta che il plasmide puc18 (che è stato usato in questo caso) abbia solo una sequenza di riconoscimento per ecoR1, e quindi dovrebbe produrre un frammento solo, giusto? Come mai in questo caso ce ne sono 2? Mi viene il dubbio che ci sia un errore nelle "etichette" delle colonne..
In realtà nel pUC18 c'è un solo sito di restrizione sia per EcoRI che per HindIII, quindi in ogni caso sarebbe strano! Qua trovi la mappa di restrizione di pUC18: sequence and restriction map
Per il logaritmo non so che dirti, bisognerebbe vedere cosa dice esattamente il testo dell'esercizio.
Comunque nel caso volessi postare cose in Inglese non ti fare assolutamente problemi! 
 |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|







