| Autore |
Discussione |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 15 novembre 2012 : 18:54:10 Inserito il - 15 novembre 2012 : 18:54:10




|
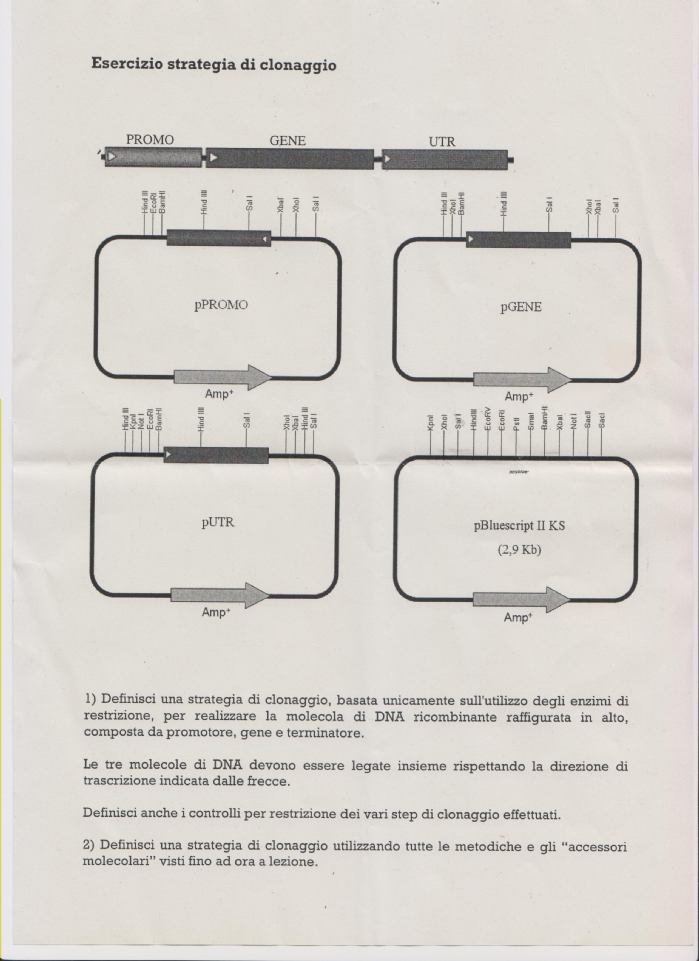
ciao a tutti i partecipanti e agli iscritti di questo forum che trovo stupendo ma soprattutto utile e didattico!
avrei un problema nel trovare la giusta strategia di clonaggio genico richiesta da questa dispensa!
potresti suggerirmi qualcosa? grazie a tutti
Immagine:
1524,56 KB
|
|
|
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 15 novembre 2012 : 19:41:12 Inserito il - 15 novembre 2012 : 19:41:12




|
| Gentilmente la prossima volta ridimensiona le immagini prima di postarle, saranno più leggere e soprattutto leggibili. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 15 novembre 2012 : 19:59:09 Inserito il - 15 novembre 2012 : 19:59:09




|
| mi dispiace mi sono iscritto oggi,non lo sapevo! grazie dell'avviso |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 16 novembre 2012 : 11:12:36 Inserito il - 16 novembre 2012 : 11:12:36




|
il prof ci ha appena detto che c'è un errore: il sito di restrizione HindIIII non esiste,è sempre HindIII.
qualche idea? |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 17 novembre 2012 : 09:42:00 Inserito il - 17 novembre 2012 : 09:42:00




|
| nessuno ha qualche idea??? |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 17 novembre 2012 : 09:48:01 Inserito il - 17 novembre 2012 : 09:48:01





|
| Beh l'idea sarebbe che tu dovresti dirci cosa ne pensi e almeno un ragionameto di massima anche se non completo e non corretto, poi se ne discute assieme. Qua non troverai nessuno che ti risolve l'esercizio, ma solo persone disposte ad aiutarti a ragionare per farti arrivare alla soluzione, quindi... la prima parola sta a te! |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 17 novembre 2012 : 09:58:21 Inserito il - 17 novembre 2012 : 09:58:21




|
allora la finalità dell'esperienza è porre nella giusta sequenza e nel giusto orientamento il promotore,il gene e la sequenza UTR.
Gene,promotore e UTR li ho in 3 vettori diversi e poi ho a disposizione un vettore vuoto che però presenta diversi siti di restrizione! La teoria degli enzimi di restrizione (ribonucleasi di classe 2) ci dice che essi catalizzano il taglio endonucleolitico del DNA per dare frammenti specifici a doppia elica con fosfati terminali al 5'.
Quindi per esempio per provocare l'escissione del promotore dal vettore numero1, lo "taglio" con Xba1 e BamH1 e per esempio lo inserisco tra gli stessi siti di restrizione presenti nel vettore vuoto! e cosi posso fare anche con gli altri! il mio problema è che non riesco a metterli nell'ordine richiesto dall'esercizio! Un 'altra cosa fondamentale è che ovviamente non si puo usare un enzima di restrizione se il sito di restrizione è presente anche all'interno del gene!
spero di essere stato chiaro! |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 20 novembre 2012 : 19:23:34 Inserito il - 20 novembre 2012 : 19:23:34




|
| nessuna illuminazione? è davvero un rompicapo! |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 20 novembre 2012 : 20:41:40 Inserito il - 20 novembre 2012 : 20:41:40




|
| si tratta solo di scegliere siti di restrizione giusti, che nn tagliano l'inserto di interesse, e che ti permettano di subclonarlo nell'orientamento giusto... |
 |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 20 novembre 2012 : 20:46:58 Inserito il - 20 novembre 2012 : 20:46:58




|
| si hai perfettamente ragione,ma credimi le ho provate tutte e non riesco a farli capitare nel giusto orientamento! cmq grazie di aver risposto |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 20 novembre 2012 : 20:55:41 Inserito il - 20 novembre 2012 : 20:55:41




|
| di nulla. L'esercizio non e' semplice, ci daro' un'occhiata. A lezione avete fatto anche tecniche come fill-in, blunting etc? |
 |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 20 novembre 2012 : 22:51:15 Inserito il - 20 novembre 2012 : 22:51:15




|
sisi certo tutte tecniche coinvolte nel clonaggio!
secondo il parere del professore questo è uno degli esempi "più semplici" figurati!
cmq grazie ancora,sei davvero gentile. |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 20 novembre 2012 : 22:53:13 Inserito il - 20 novembre 2012 : 22:53:13




|
| ah se può servire,l'aiuto che ci ha dato è di dirci che il vettore vuoto possiamo utilizzarlo anche più di una volta durante il clonaggio! |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 20 novembre 2012 : 23:26:02 Inserito il - 20 novembre 2012 : 23:26:02




|
| ok bene, ma nn capisco se il primo esercizio ti richiede di fare il clonaggio usando SOLO gli enzimi di restrizione, senza poter fare fill in, mutagenesi etc... potresti chiarire? |
 |
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 21 novembre 2012 : 13:54:29 Inserito il - 21 novembre 2012 : 13:54:29




|
| in realtà è il primo punto dell'esercizio che interessa,cioè l'utilizzo esclusivo di enzimi di restrizione per avere nel giusto orientamento quelle 3 sequenze geniche! |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 21 novembre 2012 : 14:46:32 Inserito il - 21 novembre 2012 : 14:46:32





|
Beh io un modo l'avrei trovato, non so se ce ne siano altri.
Però non voglio darti semplicemente la soluzione.
Darti un aiuto, vediamo se riesci.
1) prendi un frammento da pPROMO (non ti dico con quali enzimi) e lo inserisci nel giusto orientamento in pGENE
2) prendi un frammento da pUTR e lo inserisci in pBluescript
Ora hai due vettori che puoi mettere assieme con un altro taglio.
Prova così. |
 |
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 21 novembre 2012 : 14:55:18 Inserito il - 21 novembre 2012 : 14:55:18




|
| Anch'io avevo avuto un'idea simile, se non probabilmente la stessa. Poi pero' mi son reso conto che per uno dei due enzimi che useresti per excidere il promotore esistono due siti di restrizione in pPROMO. Ed è un peccato perché quei due siti sembravano posti in maniera strategica per permettere l'inversione del frammento. |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
gabo1991x
Nuovo Arrivato

20 Messaggi |
 Inserito il - 21 novembre 2012 : 19:25:44 Inserito il - 21 novembre 2012 : 19:25:44




|
grazie mille! cercherò di sfruttare questo consiglio :)Citazione:
Messaggio inserito da GFPina
Beh io un modo l'avrei trovato, non so se ce ne siano altri.
Però non voglio darti semplicemente la soluzione.
Darti un aiuto, vediamo se riesci.
1) prendi un frammento da pPROMO (non ti dico con quali enzimi) e lo inserisci nel giusto orientamento in pGENE
2) prendi un frammento da pUTR e lo inserisci in pBluescript
Ora hai due vettori che puoi mettere assieme con un altro taglio.
Prova così.
|
 |
|
| |
Discussione |
|






