Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
DaniBio89
Nuovo Arrivato

6 Messaggi |
 Inserito il - 19 febbraio 2013 : 19:50:46 Inserito il - 19 febbraio 2013 : 19:50:46




|
Salve a tutti, sono un bioinformatico in erba alle prese per le prime volte con R.
Sto lavorando per un progetto che consiste nel disegnare un grafico di idropatia per una determinata sequenza amminoacidica.
Se disponessi di un grafico come questo:
Immagine:

160,4 KB
Come si potrebbero colorare le aree sottese dal grafico?
Soprattutto, si possono colorare in maniera differente le aree sopra l'asse delle ascisse e quelle sotto tale asse?
Ho cercato su internet e ho trovato informazioni sulla funzione polygon(), ma non sembra fare al mio caso.
Esiste qualche pacchetto o funzione più avanzata che possa aiutarmi?
Ringrazio tutti per le eventuali risposte :)
|
|
|
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 19 febbraio 2013 : 23:20:36 Inserito il - 19 febbraio 2013 : 23:20:36




|
Io invece userei proprio polygon!
Non è proprio evidente tuttavia, devi trovare le intersezioni con l'asse x e creare poligoni multipli tra le varie intersezioni
|
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 20 febbraio 2013 : 08:00:23 Inserito il - 20 febbraio 2013 : 08:00:23




|
Un piccolo esempio in R
# Imponiamo un seed per avere un esempio riproducibile
set.seed(12345)
# Il numero di punti desiderati
num.points <- 100
# Creaimo dei valori di energia distribuiti normalmente
energy.x <- 1:num.points
energy.y <- rnorm(n=num.points, mean=0, sd=10)
# Creiamo il grafico
plot(energy.x, energy.y, t="o", pch=20, xlab="", ylab="Energia", las=1)
abline(h=0, col="darkgray", lwd=2)
# Dobbiamo trovare le intersezioni della nostra spezzata con l'asse x.
# I punti di intersezione stanno tra punti negativi e positivi
# Moltiplichiamo ciascun valore per il successivo:
# quando il segno cambia il prodotto sarà negativo
# Questa linea ci trova tutti i punti prima dell'intersezione
crossings <- energy.y[-length(energy.y)] * energy.y[-1]
crossings <- which(crossings < 0)
# Possiamo disegnarli per confema (scommentare la linea successiva)
# points(energy.x[crossings], energy.y[crossings], col="red", pch="X")
# Ora ci passiamo tutti i punti prima dell'intersezione e troviamo
# l'effettiva intersezione usando una proporzione.
# Visto che i problemi di geometria delle medie servivano a qualcosa?
intersections <- NULL
for (cr in crossings)
{
new.int <- cr + abs(energy.y[cr])/(abs(energy.y[cr])+abs(energy.y[cr+1]))
intersections <- c(intersections, new.int)
}
# Confermiamo che le intersezioni siano corrette (scommentare la linea successiva)
# points(intersections, rep(0, length(intersections)), pch=20, col="red", cex=0.7)
last.intersection <- 0
for (i in intersections)
{
ids <- which(energy.x<=i & energy.x>last.intersection)
poly.x <- c(last.intersection, energy.x[ids], i)
poly.y <- c(0, energy.y[ids], 0)
if (max(poly.y) > 0)
{
col="green"
}
else
{
col="red"
}
polygon(x=poly.x, y=poly.y, col=col)
last.intersection <- i
}
Che ci dà questo come risultato:
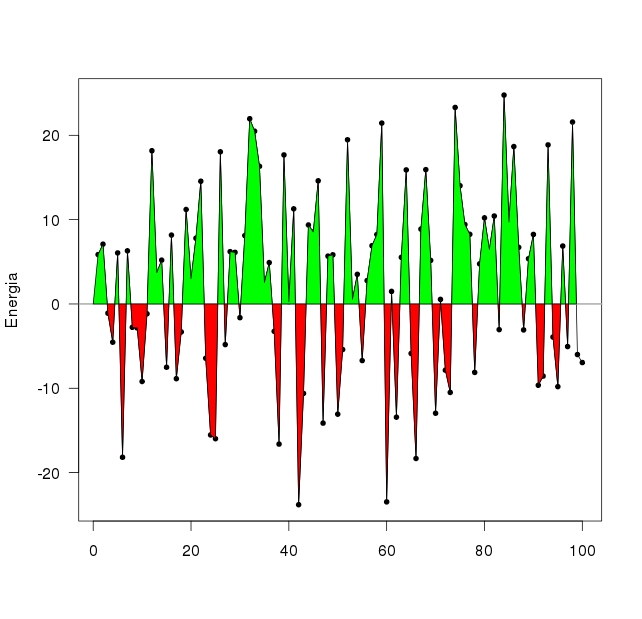 |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
DaniBio89
Nuovo Arrivato

6 Messaggi |
 Inserito il - 20 febbraio 2013 : 20:13:36 Inserito il - 20 febbraio 2013 : 20:13:36




|
Grazie per la prontezza di risposta!
Scusami se rispondo tardi ma non posso stare al pc la mattina e sono stato a studiare il tuo codice.
Come avrai capito sono un biologo che sta imparando a programmare. Ho avuto esperienze solo con il C e con Ruby. Molte funzioni di R mi sono sconosciute e il tuo codice presenta un pò di punti che non mi sono chiari al 100% (ma solo per via della mia ignoranza riguardo a R!).
Posso implementare da solo un codice, se mi fosse chiaro il motivo della "proporzione". Come mai bisogna utilizzare i due punti precedenti a ogni intersezione?
Come si imposta la proporzione?
Scusa se t faccio questa domanda di matematica magari banale, ma non riesco proprio a capire come impostare la proporzione e come mai funziona.
 |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 21 febbraio 2013 : 08:20:07 Inserito il - 21 febbraio 2013 : 08:20:07




|
In breve l'idea è questa:
Considera due punti (indicati con o nello schema qui sotto), uno sopra e uno sotto all'asse x
Ho indicato con x l'intersezione.
Se tracci due triangoli rettangoli come in figura sai che sono simili, in quanto l'angolo tra l'ipotenusa ed il cateto maggiore è lo stesso
o--|
\ |
\|
---x--------
|\
|_o
Quindi essendo simili il rapporto fra i cateti minori è uguale a quello fra i cateti maggiori.
A questo punto la riga incriminata è:
new.int <- cr + abs(energy.y[cr])/(abs(energy.y[cr])+abs(energy.y[cr+1]))
L'intersezione avrà come coordinata x la coordinata del punto (cr) più la lunghezza del cateto minore del triangolo a sinistra.
NOTA: Mi accorgo adesso che qui in realtà ho preso una scorciatoia in quanto nell'esempio i punti distano 1 unità in x... teniamo questa assunzione per il momento.
quello che faccio è prendere la coordinata y del punto a sinistra (in valore assoluto, non ci interessa a questo punto se sia positiva o negativa) e dividerla per la distanza in y tra il punto e quello successivo, ossia la somma dei due cateti maggiori.
In pratica trovo che percentuale della distanza verticale è data dal punto a sinistra.
Ad es. se y1 = 10 e y2 = -5
abs(10) / (abs(10) + abs(-5)) = 10 / 15 = 2/3
Quindi l'intersezione su x sarà a 2/3 della distanza in x tra i due punti. Assumendo che la distanza sia 1 l'intersezione sarà a cr (coordinata x del punto a sinistra) + 2/3.
Per generalizzare dobbiamo calcolare la distanza tra i due punti in x e moltiplicarla per il rapporto calcolato precedentemente.
new.int <- energy.x[cr] + abs(energy.y[cr])/(abs(energy.y[cr])+abs(energy.y[cr+1])) *
(energy.x[cr+1]-energy.x[cr])
Nota che la coordinata x è energy.x[cr] e non cr, in quanto cr è solamente l'indice della coordinata (le due cose corrispondono nel caso dell'esempio di prima, in cui i punti erano equispaziati di 1 e il primo aveva coordinata 1).
Con questa ultima correzione il tutto funziona anche con coordinate negative, non equispaziate etc. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
DaniBio89
Nuovo Arrivato

6 Messaggi |
 Inserito il - 22 febbraio 2013 : 19:26:27 Inserito il - 22 febbraio 2013 : 19:26:27




|
Grazie!
Una precisazione: ho capito che la logica di base, ovvero, la coordinata in ascissa del punto di intersezione e' pari all'ascissa del punto che precede l'intersezione sommata alla distanza esistente tra il punto di intersezione e il punto che precede l'intersezione.
Il punto che precede l'intersezione è uno dei due cateti del triangolo che si viene a "creare" dal punto più a sinistra.
Se chiamo il punto che precede l'intersezione "A" e quello che invece si trova dopo "B" allora l'ascissa del punto di intersezione "I" e':
I(x) = A(x) + cateto del triangolo A
e il cateto del triangolo A è:
cateto del triangolo A = A(y) / [A(y) + B(y)]
Non mi è chiara quest'ultima relazione.
Mi hai detto che i due triangoli sono uguali e che, quindi, il rapporto tra i cateti minori è uguale al rapporto tra i cateti maggiori. Quindi:
A cat_min : A cat_max = B cat_min : B cat_max
Il cateto del triangolo A può essere o il cateto minore di A o quello maggiore (penso che vari a seconda delle coordinate del punto).
Come si passa da questa relazione a quella che hai scritto te?
Inoltre... ho preso la tua idea e l'ho implementata in un codice scritto da me. Ma stranamente i punti di intersezione non si trovano nel punto in cui si dovrebbero trovare. Per un problema di approssimazione (credo) si trovano o leggermente a destra o leggermente a sinistra.
Ciò causa del problemi quando si usa la funzione polygon()!
Ti lascio qui un grafico:
- i punti neri rappresentano tutti i punti
- i punti rossi rappresentano quelli precedenti a una intersezione
- i punti blu quelli sull'asse delle X
Allegato:  grafico2.pdf grafico2.pdf
4,43 KB
E questo è il corrispondente grafico (con un solo colore):
Allegato:  grafico3.pdf grafico3.pdf
5,02 KB
Non capisco se è un problema di approssimazione o del mio codice.
Non capisco dove sbagli nonostante la formula che uso sia la stessa tua.
P.s. il mio modo di programmare è un pò brutale, lascio qui il codice:
# vettori che contengono le coordinate x e y dei vari punti
# le ordinate sono generate casualmente grazie alla funzione sample
vx<-c()
vy<-c()
for(i in 1:100){
vx[i]=i
vy[i]=sample(-10:10,1)
}
# il grafico viene disegnato
plot(vx,vy,main="Grafico di idropatia",xlab="Amminoacidi",ylab="Energia libera",type="l",col.axis="blue",las=1)
abline(h=0)
# ricerca di tutti i punti che precedono un'intersezione con l'asse delle x
int_x <- c()
int_y <- c()
d = 1
int_x[d]=0 # inserisco un primo punto in 0,0 (mi serve per colorare la prima area)
int_y[d]=0
for(i in 1:(100-1)){
if((vy[i]*vy[i+1]) <= 0){ # relazione da te suggeritami
d=d+1
int_x[d]=vx[i]
int_y[d]=vy[i]
}
}
int_x[d+1]=vx[100]
int_y[d+1]=0
points(int_x,int_y,col="red",pch=20)
# ricerca di tutte le intersezioni sull'asse delle x
num_int = length(int_x) #ci si può mettere anche int_y
int_pol_x <- c()
int_pol_y <- c()
for(i in 1:num_int){
int_pol_x[i] = ( int_x[i] + ( abs(int_y[i])/( abs(int_y[i])+abs(int_y[i+1]) ) ) )
int_pol_y[i] = 0
}
points(int_pol_x,int_pol_y,col="blue",pch=20)
Scusa se ti continuo a chiedere queste cose ma non capisco dove sbaglio, sono due giorni che ci sbatto la testa... |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 23 febbraio 2013 : 10:02:12 Inserito il - 23 febbraio 2013 : 10:02:12




|
Attenzione, i due triangoli sono SIMILI, non uguali. Uno è la versione "più piccola" dell'altro.
Comunque, è un po' complesso disegnarlo qui e non ho molto tempo adesso... ma l'idea è che puoi costruire un terzo triangolo che ha come cateti la somma dei cateti degli altri due e dimostrare che anche lui è simile agli altri due. Dopodichè scrivi la proporzione fra i lati di questo "triangolone" e quelli di un altro dei triangoli più piccoli ed ottieni la mia formula (la seconda versione, quella corretta).
Sinceramente non vedo dove stia il problema nel codice... deve essere un problema di arrotondamento, prova a far scrivere (usando print) i vari risultati mano a mano che il codice procede. Magari usa 10 punti invece di 100 così non hai milioni di valori da controllare.
PS: nota che
vx<-c()
vy<-c()
for(i in 1:100){
vx[i]=i
vy[i]=sample(-10:10,1)
}
può essere semplicemente sostituito da
vx = 1:100
vy = sample(-10:10, 100, replace=TRUE)
O, se non vuoi restringerti a coordinate intere
vy = sample(seq(-10,10,0.01), 100, replace=TRUE)
|
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
DaniBio89
Nuovo Arrivato

6 Messaggi |
 Inserito il - 10 marzo 2013 : 17:05:41 Inserito il - 10 marzo 2013 : 17:05:41




|
Scusa se non ti ho ho più risposto ma in queste due settimane sono stato un po' occupato.
Adesso il codice funziona (avevo commesso un errore di cui non mi ero accorto).
Ti ringrazio tantissimo per l'aiuto che mi hai dato e per avermi chiarito i vari dubbi.
Grazie 1000! |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|







