Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
carlo4567
Nuovo Arrivato
Prov.: roma
Città: roma

40 Messaggi |
 Inserito il - 23 settembre 2005 : 15:56:51 Inserito il - 23 settembre 2005 : 15:56:51




|
Vorrei trovare il sito catalitico della protein kinase umana.
Credo che si faccia con PROSITE, ma non ho chiaro il procedimento.
Qualcuno può darmi un'aiuto?
Grazie
|
|
|
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 24 settembre 2005 : 02:28:35 Inserito il - 24 settembre 2005 : 02:28:35




|
Quale protein kinasi? Ce ne sono centinaia!
Non ho mai usato PROSITE, ma ho fatto una veloce prova (con la protein kinasi A (PKA))
Fai una ricerca di : protein kinase
Non ti trovera' un match esatto, ma l'ultima entry e'
PDOC00004 cAMP- and cGMP-dependent protein kinase phosphorylation site
(PKA e' cAMP dipendente).
Troverai che il sito che fosforila (sul substrato) e':
[RK](2) - x - [ST] (S or T is the phosphorylation site)
Non sono pero' riuscito a trovare il sito attivo... quindi sono andato su RCSB (www.rcsb.org) che a mio parere e' molto meglio. 
Quindi su RCSB cerchi cAMP dependent protein kinase (sembra che PKA non piaccia a 'sta gente...)
e ti trova un sacco di entry.
Io ho scelto la seconda (1ATP) che e' la struttura del dominio catalitico.
Ora, nella pagina che ti viene fuori puoi vedere tutti i vari dettagli della proteina, divertiti un po' a esplorarlo, questo db e' fatto proprio bene.
Ora, nel menu a sx scegli Geometry e nella pagina che ti carica scegli "Motif summary (PROMOTIF)".
Si aprira' una nuova finestra con la sequenza della proteina, che ti mostra 2 motivi.
Domain Links CATH no. Class Architecture
1 (CATH) (DHS) 1.10.510.10 = Mainly Alpha Orthogonal Bundle
2 (CATH) (DHS) 3.30.200.20 = Alpha Beta 2-Layer Sandwich
Cliccando su CATH ti fara' vedere le due sequenze dei domini catalitici.
In piu', se nella barra in alto scegli "Ligands" ti fa vedere il sito catalitico con legata l'ATP.
Se cerchi un po' in giro su Google, tra l'altro, troverai sicuramente l'esatto meccanismo di reazione!
Buona ricerca!!!
nICO |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
carlo4567
Nuovo Arrivato
Prov.: roma
Città: roma

40 Messaggi |
 Inserito il - 24 settembre 2005 : 08:19:44 Inserito il - 24 settembre 2005 : 08:19:44




|
Hai ragionesono stato impreciso e per di più ho sbagliato a scrivere
In realtà nella fretta ho sbagliato a scrivere.
La domanda é
Dove si trova il sito catalitico della Piruvato kinasi nella sequenza il cui codice di accesso a SwissProt è P14618?
Ti spiego come ho fatto.
Sono andato al sito
http://ca.expasy.org/
e ho cercato il codice P14618 in swissprot
Ho trasformato la sequenza in formato FASTA
sempre da expasy.org ho scelto prosite in alto a sx
Nella pagina che si apre , in basso c'è una text area, dentro la quale ho incollato la sequenza.
Qundi ho cliccato su quick scan
Ha questo punto mi trovo in difficoltà.
nella pagina dei risultati (http://ca.expasy.org/cgi-bin/prosite/ScanView.cgi?scanfile=30253597111.scan.gz)
penso che il sito catalitico sia
PS00005 PKC_PHOSPHO_SITE Protein kinase C phosphorylation site :
40 - 42: TaR
59 - 61: TlK
86 - 88: TiK
138 - 140: TlK
204 - 206: SkK
221 - 223: SeK
364 - 366: TaK
419 - 421: SfK
433 - 435: SgR
458 - 460: TaR
523 - 525: TmR
Come faccio però a interpetare i risultati?
Quale é la risposta alla domanda dove si trova il sito catalitico?
Grazie per ogni suggerimento
Citazione:
Messaggio inserito da chick80
Quale protein kinasi? Ce ne sono centinaia!
Non ho mai usato PROSITE, ma ho fatto una veloce prova (con la protein kinasi A (PKA))
Fai una ricerca di : protein kinase
Non ti trovera' un match esatto, ma l'ultima entry e'
PDOC00004 cAMP- and cGMP-dependent protein kinase phosphorylation site
(PKA e' cAMP dipendente).
Troverai che il sito che fosforila (sul substrato) e':
[RK](2) - x - [ST] (S or T is the phosphorylation site)
Non sono pero' riuscito a trovare il sito attivo... quindi sono andato su RCSB (www.rcsb.org) che a mio parere e' molto meglio. 
Quindi su RCSB cerchi cAMP dependent protein kinase (sembra che PKA non piaccia a 'sta gente...)
e ti trova un sacco di entry.
Io ho scelto la seconda (1ATP) che e' la struttura del dominio catalitico.
Ora, nella pagina che ti viene fuori puoi vedere tutti i vari dettagli della proteina, divertiti un po' a esplorarlo, questo db e' fatto proprio bene.
Ora, nel menu a sx scegli Geometry e nella pagina che ti carica scegli "Motif summary (PROMOTIF)".
Si aprira' una nuova finestra con la sequenza della proteina, che ti mostra 2 motivi.
Domain Links CATH no. Class Architecture
1 (CATH) (DHS) 1.10.510.10 = Mainly Alpha Orthogonal Bundle
2 (CATH) (DHS) 3.30.200.20 = Alpha Beta 2-Layer Sandwich
Cliccando su CATH ti fara' vedere le due sequenze dei domini catalitici.
In piu', se nella barra in alto scegli "Ligands" ti fa vedere il sito catalitico con legata l'ATP.
Se cerchi un po' in giro su Google, tra l'altro, troverai sicuramente l'esatto meccanismo di reazione!
Buona ricerca!!!
nICO
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 25 settembre 2005 : 02:21:38 Inserito il - 25 settembre 2005 : 02:21:38




|
Citazione:
penso che il sito catalitico sia
PS00005 PKC_PHOSPHO_SITE Protein kinase C phosphorylation site :
No, quello e' un sito sulla piruvato chinasi che puo' essere TARGET di una fosforilazione da parte della protein chinasi C (PKC).
Se guardi in alto troverai
Hits by PS00110 PYRUVATE_KINASE Pyruvate kinase active site signature :
sp-P14618-KPY~ sp-P14618-KPYM_HUMAN hits (530 aa)
264 - 276: IkIISKIENhEGV
Questo e' il sito attivo!
Se vuoi vedere dove sta puoi andare su www.rcsb.org e scaricare la struttura da guardare con RasMol o il programma che preferisci o direttamente guardarla online.
Ti allego una veloce immagine del sito attivo fatta con Rasmol.
Enjoy
nICO
Immagine:
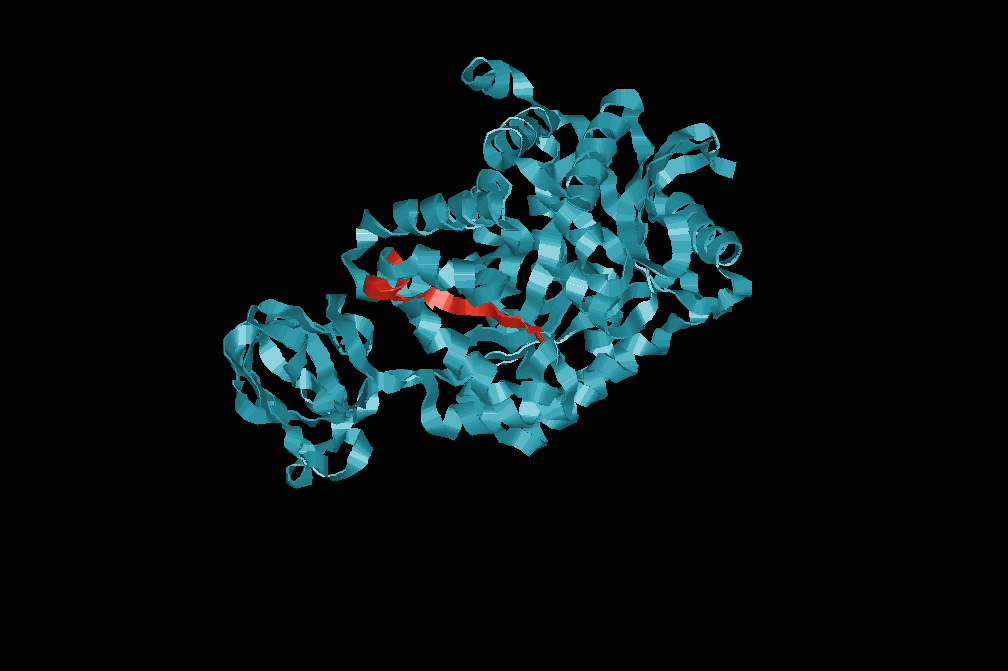
33,44 KB |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|




