| Autore |
Discussione |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 20 agosto 2009 : 15:14:15 Inserito il - 20 agosto 2009 : 15:14:15




|
Salve a tutti coloro i quali sono ancora a lavoro.
Recentemente lavorando sull’mRNA di p75 andando ad indagare sulla presenza dei 6 esoni, in prima analisi, utilizzando dei primer esterni per il primo questo non veniva amplificato per cui si poteva presupporre uno spicing.
Indagando ulteriormente ed utilizzando due primer foward più interni (uno a partire dal nucleotide 64 e l’altro dal nucleotide 84) e come revers il revers del secondo esone, dal 64 non amplificava mentre dall’84 si.
Cosa pensate possa essere successo????
Questa condizione di mancanza dei primi nucleotidi può condurre ad un modificazione del frame di lettura nella traduzione per cui la proteina viene tradotta ma in modo diverso per cui assume caratteristiche diverse da quelle naturali?????
Tenete conto che al microscopio a fluorescenza la vedo in regione reticolare ma non sulla membrana.
Può essere che venga tradotta senza la regione deputata alla glicosilazione, per cui non si innescano i meccanismi di trasporto alla membrana?????
http://www.ncbi.nlm.nih.gov/nuccore/NM_002507.2?report=genbank
Studiando su questo link è possibile ricavare come entrano le triplette del codon per arrivare al CDS (126-1409), cioè qual'è la chiave di lettura?????
Scusate per la terminologia.
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 24 agosto 2009 : 18:13:05 Inserito il - 24 agosto 2009 : 18:13:05





|
Questa è la sequenza del tuo mRNA con evidenziati i vari punti:
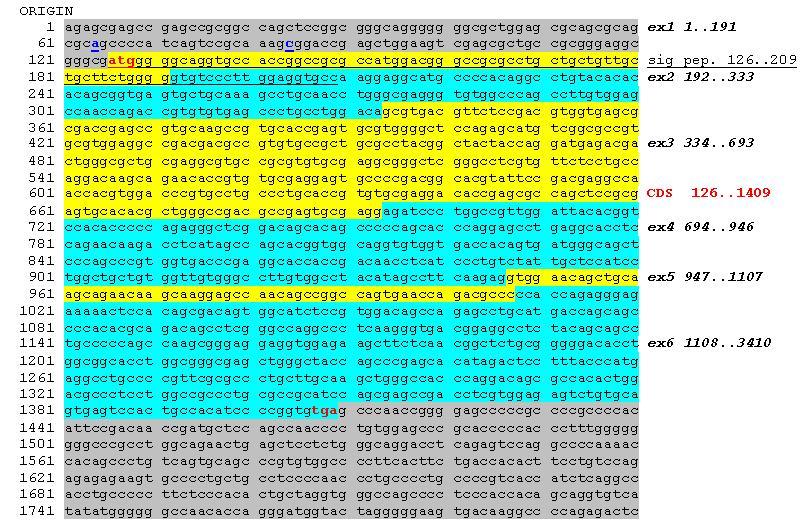
La parte che ti "dovrebbe" mancare sta nella 5'UTR (evidenziata in grigio), (ho scritto in blu i nucleotidi 64 e 84) non fa cambiare il frame di lettura perché l'ATG di inizio è a 126 (scritto in rosso).
Inoltre: Citazione:
Tenete conto che al microscopio a fluorescenza la vedo in regione reticolare ma non sulla membrana.
Strano perché la sequenza segnale c'è (a quanto pare) è quella da 126 a 209 (che ho sottolineato), magari dovresti controllare se è quella o è diversa.
Citazione:
Studiando su questo link è possibile ricavare come entrano le triplette del codon per arrivare al CDS (126-1409), cioè qual'è la chiave di lettura?????
Beh si, parti dalla A dell'ATG che sta a 126.
Se ti manca un pezzo della sequenza 5'UTR quello che potrebbe essere influenzato è il legame del ribosoma o di fattori di inizio della traduzione o altri fattori regolatori, fatto sta che comunque la proteina se la vedi viene tradotta.
 |
 |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 25 agosto 2009 : 18:20:53 Inserito il - 25 agosto 2009 : 18:20:53




|
Immagine:

73,71 KB
quale dei tre eventi è il giusto frame di lettura.
E poi: mancando una parte del 5’UTR, l’ingresso anomalo nel ribosoma può alterare il frame di lettura e far si che l’AUG in posizione 126 venga saltato e la traduzione inizi più a valle?
|
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 25 agosto 2009 : 18:44:21 Inserito il - 25 agosto 2009 : 18:44:21





|
  
Il frame di lettura inizia sempre da ATG!!!
Quindi sarà sempre:
ATG tut toq uel llo che vie ned opo!! *
Gli eventi 1 e 3 sono impossibili!!!
(* N.B. per i lettori più attenti l'inserzione di base non è casuale, è voluta per agevolare la lettura!) |
 |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 25 agosto 2009 : 23:38:39 Inserito il - 25 agosto 2009 : 23:38:39




|
Grazie per avermi confermato l'evento 2 
e per la seconda domanda, cosa ne pensate 
Proverò, comunque, quando avrò i fondi, a immunoprecipitare e testare se la proteina è glicosilata. |
 |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 26 agosto 2009 : 10:09:54 Inserito il - 26 agosto 2009 : 10:09:54




|
chick80 vedo che sei in linea.
tu cosa ne pensi  |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 26 agosto 2009 : 15:19:46 Inserito il - 26 agosto 2009 : 15:19:46




|
Concordo con Silvia. La cosa più facile sarebbe sequenziare la sequenza per vedere se ci sono strane mutazioni?
La localizzazione la fai al confocale o con un microscopio a epifluorescenza? |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 26 agosto 2009 : 18:09:08 Inserito il - 26 agosto 2009 : 18:09:08




|
la localizzazione è al microscopio a epifluorescenza.
ma ditemi!!! come faccio a sequenziare qualcosa che non mi si amplifica? quali primer dovrei usare? sono in alto mare.
sulla glicosilazione, cosa mi dite?
grazie ragazzi |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 26 agosto 2009 : 19:45:27 Inserito il - 26 agosto 2009 : 19:45:27





|
Allora cerchiamo di fare un attimo di chiarezza:
Primo: il Sequenziamento
Citazione:
Messaggio inserito da farmacologia
come faccio a sequenziare qualcosa che non mi si amplifica?
Citazione:
Messaggio inserito da farmacologia
Indagando ulteriormente ed utilizzando due primer foward più interni (uno a partire dal nucleotide 64 e l'altro dal nucleotide 84) e come revers il revers del secondo esone, dal 64 non amplificava mentre dall'84 si.
Questo hai detto che si amplifica, quindi puoi sequenziarlo e sarebbe già un punto da cui partire!
Per sequenziarlo puoi benissimo utilizzare gli stessi primers che hai utilizzato per la PCR.
Poi potresti pensare di fare una analisi migliore della tua estremità 5' utilizzando una RACE PCR . Cosa non proprio facilissima!
Secondo: la sequenza segnale
è la sequenza segnale che è importante per il trasporto delle proteine nelle varie localizzazioni.
Ti consiglio qualche lettura in proposito, ad es. dall'Alberts:
Signal Sequences and Signal Patches Direct Proteins to the Correct Cellular Address
Quella come ti dicevo, sembra esserci:
Citazione:
Messaggio inserito da GFPina
Strano perché la sequenza segnale c'è (a quanto pare) è quella da 126 a 209 (che ho sottolineato), magari dovresti controllare se è quella o è diversa.
Ma questo dovresti controllarlo appunto "sequenziando" il tuo frammento!
Terzo: la Glicosilazione
Innanzitutto dovresti sapere "dove" è glicosilata la tua proteina! E controllare se l'aminoacido/i che viene/vengono glicosilati sono presenti.
Citazione:
Messaggio inserito da farmacologia
Proverò, comunque, quando avrò i fondi, a immunoprecipitare e testare se la proteina è glicosilata.
BTW basta un Western Blot!
Dal link di UNIPROT sembrerebbe che la tua proteina sia glicosilata all'aminoacido 60: http://www.uniprot.org/uniprot/P08138
ti linka anche questo articolo: O-linked oligosaccharide on the 75-kDa neurotrophin receptor.
magari ti può essere utile.
Premetto che io non l'ho letto e non so cosa faccia la glicosilazione alla tua proteina.
Mi sembra di aver risposto a tutto questa volta, nel caso dimmi se c'è qualche punto ancora oscuro.
  |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 26 agosto 2009 : 19:53:45 Inserito il - 26 agosto 2009 : 19:53:45




|
Citazione:
la localizzazione è al microscopio a epifluorescenza.
Quindi non puoi essere sicuro al 100% che non sia presente in membrana... io un'occhiata al confocale la darei, ci vuole un attimo.
EDIT: Silvia mi fa notare che probabilmente stai usando delle cellule in coltura... in quel caso l'epifluorescenza può andare bene. Su fettine di tessuto mi fiderei di meno |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 27 agosto 2009 : 09:02:41 Inserito il - 27 agosto 2009 : 09:02:41




|
si, sono cellule in coltura.
comunque farò tesoro dei vostri consigli.
se vi viene in mente qualcos'altro, tenetemi in considerazione.
grazie ragazzi, vi farò sapere.  |
 |
|
| |
Discussione |
|







