| Autore |
Discussione |
|
|
Moloney
Nuovo Arrivato

111 Messaggi |
 Inserito il - 09 maggio 2009 : 20:22:28 Inserito il - 09 maggio 2009 : 20:22:28




|
Ciao a tutti!
Sto cercando di adottare una strategia per lo studio dei trascritti alternativi di un gene, usp14 di topo, da ensembl. Qui il link
http://www.ensembl.org/Mus_musculus/Gene/Splice?g=ENSMUSG00000047879
Voglio usare una RT-PCR, ma non sono riuscito a trovare 1 coppia ( o 2 coppie) di primer che mi permettessero di discriminare (senza guardare lo spessore della banda) tra i 3 trascritti. Se non trovate niente di meglio, vi dico cosa avevo pensato io.
Grazie!
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 10 maggio 2009 : 09:18:44 Inserito il - 10 maggio 2009 : 09:18:44





|
Ciao!
Non ho controllato di cosa si tratta, ma hai provato a disegnare i primers con Primer Blast?
Dovresti vedere se sono specifichi per i trascritti alternativi.
 |
 |
|
|
Moloney
Nuovo Arrivato

111 Messaggi |
 Inserito il - 10 maggio 2009 : 12:14:08 Inserito il - 10 maggio 2009 : 12:14:08




|
| Ciao GFPina! Il programma che uso è primer3 e il problema non è disegnare i primers, ma adottare una strategia che mi permetta di discernere i 3 trascritti! Il programma che mi hai suggerito non mi consente ciò! (anche se non lo conoscevo e mi sembra ottimo!) |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 10 maggio 2009 : 12:52:10 Inserito il - 10 maggio 2009 : 12:52:10




|
Ho dato solo un'occhiata veloce... ma direi che sicuramente USP14-001 è facile da determinare, in quanto ha un esone in più (ENSMUSE00000140483)
Le altre 2 varianti sono diverse principalmente al 5' e al 3'.
Potresti quindi avere 2 coppie di primers, una attorno a quell'esone extra, che quindi ti darà una banda + lunga per il trascritto 001 e più corta con gli altri 2 trascritti. Poi un'altra coppia di primer che potrebbe essere specifica per l'inizio del trascritto 201 e non amplificare nulla sugli altri 2.
Quindi:
trascritto 001 -> primers 1: 1 banda + lunga
trascritto 002 -> primers 1: 1 banda + corta
trascritto 201 -> primers 1: 1 banda + corta e primers 2: 1 banda |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
Moloney
Nuovo Arrivato

111 Messaggi |
 Inserito il - 10 maggio 2009 : 12:59:12 Inserito il - 10 maggio 2009 : 12:59:12




|
hai pensato la mia stessa cosa! Il problema è che potrei dover detectare in un tessuto più isoforme, ad esempio la 1 e la 3; la 2 e la 3.
Ora ti copincollo quello che avevo pensato io.
con prima coppia di primers fra esone 3 e 5, distinguo fra 1° trascritto e gli altri 2.
Con seconda coppia di primers sul terminale UTR 3’ se c’è banda, corrisponde al trascritto 3. Se non c’è banda, avrò solo il trascritto 1 0 2 o 1 E 2. Così:
Se ho 2 bande nella prima corsa, ho trascritto 1 e trascritto 2 o 3 o 2E3.
Se ho 1 banda nella prima corsa: NON ho trascritto 1 e posso avere trascritti 2 o 3 o 2E3.
ho trascritto 1 e NON ho trascritti 2 o 3 o 2E3.
Se non ho bande nella seconda corsa, ho trascritti 1 e 2, ma NON ho il 3.
Se ho 1 banda, HO trascritto 3.
Trascritto 1: banda 1 in prima corsa; nessuna banda in seconda corsa
Trascritto 2: banda 2 in prima corsa; nessuna banda in seconda corsa
Trascritto 3: banda 2 in prima corsa; 1 banda in seconda corsa
Trascritto 1- 3: banda 1 e 2 in prima corsa; 1 banda in seconda corsa
Trascritto 2-3: banda 2 in prima corsa; 1 banda in seconda corsa.
Trascritto 1-2: banda 1 e 2 in prima corsa; nessuna banda in seconda corsa
Trascritto 1-2-3: banda 1 e 2 in prima corsa; 1 banda in seconda corsa.
Nei casi messi in grassetto, si ha lo stesso tipo di profilo, quindi non posso discernere.. |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 10 maggio 2009 : 15:53:29 Inserito il - 10 maggio 2009 : 15:53:29





|
Citazione:
Messaggio inserito da Moloney
Ciao GFPina! Il programma che uso è primer3 e il problema non è disegnare i primers, ma adottare una strategia che mi permetta di discernere i 3 trascritti! Il programma che mi hai suggerito non mi consente ciò! (anche se non lo conoscevo e mi sembra ottimo!)
Il punto è proprio quello, a patto che le 3 forme alternative dei trascritti siano presenti nel database di NCBI (non ho controllato però) ti consente proprio di discriminare! E' diverso da altri programmi per disegnare primers perchè fa anche il blast e cerca primers specifici solo per quel trascritto e nel caso i primers riconoscano anche un altro trascritto o una diversa variante di splicing te lo segnala.
L'unico inconveniente è appunto che fa il blast sulle sequenze inserite nei database di NCBI, quindi se non sono inserite non permette questa discriminazione.
Scusa ma non ho letto ancora con attenzione quello che hai scritto!
Per ora ti rispondo solo questo...
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 10 maggio 2009 : 15:55:49 Inserito il - 10 maggio 2009 : 15:55:49




|
Ok, ho capito il problema... non ho provato a disegnare veramente i primers, ma con un approccio a 5 primers come in figura dovrebbe funzionare un po' meglio.
Le distanze fra i primer sono messe un po' a caso... Inoltre nel mio disegno ipotizzo che tu possa fare la PCR con tutti e 5 i primers insieme, ma non è detto che in pratica sia veramente fattibile (anche se si potrebbe provare).
Le uniche situazioni non distinguibili sono 1+2+3 e 1+3 ma sinceramente non mi vengono in mente altre soluzioni al momento
Immagine:
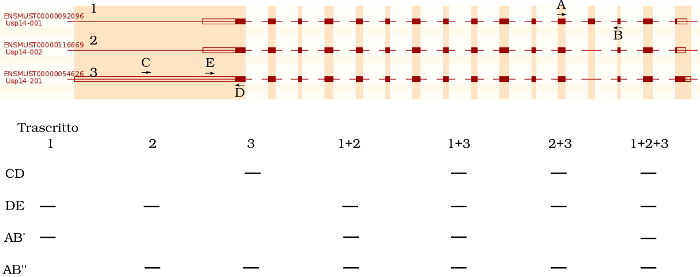
71,9 KB |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 10 maggio 2009 : 15:57:26 Inserito il - 10 maggio 2009 : 15:57:26




|
| PS: non ho voglia di rifare il disegno... ma sarebbe meglio avere D sull'esone successivo, così ti eviti anche problemi da contaminazione da DNA genomico |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
Moloney
Nuovo Arrivato

111 Messaggi |
 Inserito il - 10 maggio 2009 : 19:06:21 Inserito il - 10 maggio 2009 : 19:06:21




|
| penso che l' unica sia fare un 3' RACE e sequenziamento... |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 10 maggio 2009 : 19:59:57 Inserito il - 10 maggio 2009 : 19:59:57





|
Si penso che il sequenziamento possa essere un'idea.
Ho controllato le sequenze e il fatto è che la variante 2 è perfettamente identica alla 3 a parte che la 3 ha dei pezzi in più a 3' e 5', quindi puoi disegnare primers specifici per la 1 e per la 3, ma non puoi disegnare nessun primer specifico per la 2, perchè qualunque coppia di primers disegni riconoscerà anche la 3.
Io ho fatto una modifica al disegno di chick80 (visto che era già bello fatto  ), ma è più o meno quello che dicevi tu, l'unica variante è che io disegnerei i primers a cavallo degli esoni, in questo modo discrimini tra i trascritti per presenza/assenza di banda e non per la lunghezza delle due bande e eviti qualsiasi contaminazione da DNA genomico, i primers non si legheranno neanche al DNA. ), ma è più o meno quello che dicevi tu, l'unica variante è che io disegnerei i primers a cavallo degli esoni, in questo modo discrimini tra i trascritti per presenza/assenza di banda e non per la lunghezza delle due bande e eviti qualsiasi contaminazione da DNA genomico, i primers non si legheranno neanche al DNA.
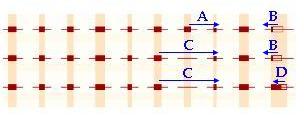
- la coppia di primer AB riconosce solo il trascritto 1
- la coppia di primer CB riconosce sia il trascritto 2 che il 3
- la coppia di primer CD riconosce solo il trascritto 3
altra cosa, ma non affidabilissima sarebbe guardare la "quantità" relativa dei trascritti, una volta che amplifichi e ottieni la banda CD (che può essere sia del trascritto 2 che 3), se dalla PCR CD non ottieni amplificato sei sicuro che ci sia solo il 2 e sei a posto. Se invece hai amplificato non sai se è dovuto solo al 3 o se c'è anche il 2. A questo punto quantificando: se la quantità di amplificato CD (trascritto 3) è uguale a quella dell'amplificato CB (trascritto 2 o 3) allora è verosimile tu abbia solo il 3, se invece è inferiore hai anche il 2. Certo non è un metodo molto accurato.
Altrimenti non ti rimane che il Northern! A quel punto discrimini in base al peso. (beh almeno però puoi fare una prima analisi per PCR e fare il Northern solo sui campioni dubbi)
Altro non mi viene in mente!
 |
 |
|
|
Moloney
Nuovo Arrivato

111 Messaggi |
 Inserito il - 10 maggio 2009 : 23:24:21 Inserito il - 10 maggio 2009 : 23:24:21




|
si, penso che alla fine la RT-PCR non mi darà sicurezze nei campioni con 2-3, quindi o RACE e sequenziamento o northern se ho bande dubbie, anche perchè della quantificazione delle bande mi fido pochino! Grazie a tutti! |
 |
|
| |
Discussione |
|







