Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
STEFANIApcr
Nuovo Arrivato

Prov.: Brescia
Città: Brescia

26 Messaggi |
 Inserito il - 14 febbraio 2013 : 12:31:29 Inserito il - 14 febbraio 2013 : 12:31:29




|
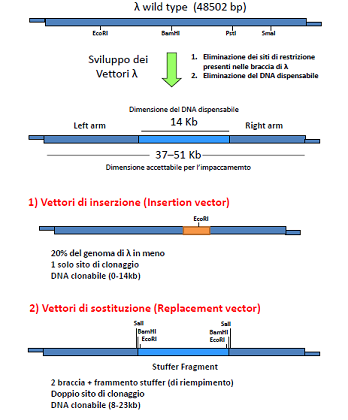
Ciao ragazzi, ho un problema per quanto riguarda la costruzione di vettori fagici utilizzati nelle tecniche di clonaggio. ciò che non riesco a capire è la differenza tra frammento di riempimento e frammento stuffer. quale dei due è la regione non indispensabile per il ciclo litico di lambda? come sono stati costruiti i vettori di inserzione e di sostituzione? ho capito che, il vettore di inserzione contiene un sito unico di restrizione in uno dei geni della lisogenia (ad esempio nel gene per il repressore cI), ma: il sito di restrizione è già presente nella regione non indispensabile o è stato messo artificialmente? e in tal caso come è stato costruito?
per quanto riguarda il vettore di sostituzione, questo ha una regione con siti di restrizione ai lati di un frammento:stuffer? riempimento? regione non indispensabile?
vi ringrazio anticipatamente!!  
Immagine:
46,76 KB
|
|
|
|
|
Uther
Nuovo Arrivato

Prov.: Napoli
Città: Na

35 Messaggi |
 Inserito il - 15 febbraio 2013 : 13:44:53 Inserito il - 15 febbraio 2013 : 13:44:53




|
Ciao =) Partiamo dal fatto che il DNA del fago lambda selvatico (per esempio)contiene molti siti per enzimi di restrizione che sono normalmente impiegati in biologia molecolare, ad esempio Sau3AI (che tra l'altro è a taglio frequente quindi è comodo). Quindi non hai bisogno di modificare il genoma del fago su cui stai lavorando per inserire siti di restrizione; ti basta avere una mappa del suo genoma dove sono riportati i siti di restrizione e in base a quello che devi fare scegliere i siti più consoni.
Ad esempio puoi sfruttare il sito BamHI (presente nella posizione X) per inserire un frammento di DNA esogeno che precedentemente hai tagliato con BamHI; in questo modo allestisci un vettore di INSERZIONE, in quanto inserisci un frammento genico senza eliminare nessuna sequenza virale.Ovviamente puoi inserire un gene relativamente piccolo, in quanto il capside di lambda può contenere solo il 5% di DNA in più rispetto al wt. Per tale motivo sono stati allestiti dei vettori di SOSTITUZIONE nei quali rimuovi una regione genica virale che non pregiudica l'infettività del virus, e la sostituisci con un gene esogeno completo o comunque un frammento più grande rispetto a quello che potresti inserire in un vettore di inserzione.
Vettori relativamente recenti (sono stati proposti nel 1983) come EMBL3 o EMBL4 consentono di inserire da 9 kb a 23 kb di DNA esogeno.
Il DNA stuffer è il frammento di DNA virale che rimuovi e che sostituisci; in genere lo rimuovi o per taglio enzimatico (immagina due siti di restrizione, uno a sx e uno a dx dello stuffer) o con metodi fisici (poco usato però).
Per quanto riguarda qual'è la regione non indispensabile che può essere sostituita (lo stuffer per intenderci), non ti saprei dire esattamente qual'è in lambda; non credo si tratti del gene del repressore CI perchè la "scelta" di ciclo lisogenico o litico in lambda dipende da una serie di segnali ambientali tra cui temperatura, molteplicità di infezione, disponibilità nutrizionale e fa rifermento alle concentrazioni di CI e Cro (attivatore ciclo litico)più che alla assenza o presenza. Quindi il gene repressore di lambda che favorisce il ciclo lisogenico (che sembra quindi non servire in una infezione)deve essere presente. Ad ogni modo la cosa importante è capire il concetto che c'è dietro, più che il gene specifico.
Ho cercato di essere quanto più generale possibile, per non andare fuori domanda, però se vuoi dettagli fammi sapere =P |
 |
|
|
Geeko
Utente
 
Città: Milano

1043 Messaggi |
 Inserito il - 15 febbraio 2013 : 15:03:41 Inserito il - 15 febbraio 2013 : 15:03:41




|
Comunque i vettori di sostituzione non sono semplici genomi di lambda. Mancano di una porzione centrale non indispensabile per il ciclo d'infezione del virus e presentano come dicevi un unico sito di restrizione che cade ad esempio all'interno del gene cI. Dopo l'inserzione quindi puoi individuare i fagi ricombinanti per la loro incapacità di lisogenizzare, ma di dare invece placche di lisi.
Per questi vettori non esiste una dimensione minima dell'inserto da clonare, infatti già di per sé hanno dimensioni tali da poter essere impacchettati all'interno del capside virale.
Nei vettori di sostituzione il discorso è leggermente diverso: vengono venduti nella forma comprendente il braccio sinistro - regione stuffer - braccio destro (in questa forma possono essere propagati). Tra la regione stuffer e le due braccia si trovano siti multipli di clonaggio. Dopo aver digerito il vettore con appropriati enzimi di restrizione è necessario recuperare le due braccia dopo separazione su gel di agarosio e passare a clonare l'inserto tagliato con gli stessi enzimi di restrizione. In questo caso si possono clonare inserti più grandi (fino a 23kb) ma devono comunque avere una dimensione minima di 9kb, altrimenti il costrutto è troppo corto per essere incorporato nella testa del fago (in questo caso sai per certo che le placche che ottieni derivano da un vettore ricombinante perché i vettori vuoti non funzionano).
La regione eliminata corrisponde alla porzione del genoma adibita a dirigere la lisogenia. |

|
 |
|
|
STEFANIApcr
Nuovo Arrivato

Prov.: Brescia
Città: Brescia

26 Messaggi |
 Inserito il - 16 febbraio 2013 : 12:07:29 Inserito il - 16 febbraio 2013 : 12:07:29




|
Grazie ragazzi! mi avete chiarito le idee!!! Potrei chiedervi ancora una delucidazione? Con quale meccanismo il fago lambda sfugge all'azione della esonucleasi V di coli quando, a causa dell'inserimento del Dna esogeno, viene distrutto il gene gam (codificante una proteina che interferisce l'azione dell'enzima batterico) presente nel frammento stuffer? la parte che non ho ben capito riguarda, in particolare il ruolo della proteina Red di lambda, nella formazione del dimero prima della replicazione a circolo rotante.
Grazie davvero!! |
 |
|
|
Uther
Nuovo Arrivato

Prov.: Napoli
Città: Na

35 Messaggi |
 Inserito il - 16 febbraio 2013 : 12:35:04 Inserito il - 16 febbraio 2013 : 12:35:04




|
Stai lavorando su un ceppo di Coli caratterizzato dal fenotipo Spi-, cioè è lisogeno per il fago P2; lambda selvatico è invece Spi+, non è quindi in grado di infettare batteri Spi-; è stato dimostrato che questa incapacità è dovuta ai geni red e gam. Se allestisci un vettore in cui la regione stuffer (che quindi rimuovi per inserire il frammento di DNA esogeno) è preparata in modo tale da comprendere i geni gam e red, i fagi ricombinanti (in cui è avvenuta la sostituzione) non presenteranno più i geni gam e red, diventano quindi Spi- e quindi possono infettare ceppi di E.coli lisogeni per P2. Puoi selezionare i ceppi di lambda ricombinanti proprio osservando l'infezione in ceppi di coli P2 che normalmente lambda wt non infetta.
Però la mancanza di gam e red ha effetti abbastanza forti sulla vitalità del fago; il prodotto di gam infatti è necessario sia per la replicazione del DNA bidirezionale sia per quella a circolo rotante. I fagi gam- non fomrmano i concatameri di DNA che vengono impaccati.
La possibilità di formare multimeri di DNA circolare è connessa all'attività di sistemi di ricombinazione rec/red; ma poichè manca anche red nei fagi Spi- la capacità di formare placche di lisi dipende solo da rec.
E infatti le placche di lisi, in genere, sono molto piccole perchè il DNA di lambda non è granchè come substrato del prodotto di rec, perchè manca della sequenza chi (seq. di stimolazione del crossing over).
Alcuni vettori di sostituzione infatti sono stati modificati inserendo nella parte di DNA non rimovibile un sito chi.
Per l'endonucleasi V in realtà è il prodotto di gam che inibisce il sistema RecBCD nuclease dell'ospite che degrada le estremità 3' dei circoli rotanti. |
 |
|
|
STEFANIApcr
Nuovo Arrivato

Prov.: Brescia
Città: Brescia

26 Messaggi |
 Inserito il - 17 febbraio 2013 : 19:26:56 Inserito il - 17 febbraio 2013 : 19:26:56




|
Sei stato chiarissimo! quindi, se ho capito bene: nei vettori, di sostituzione in cui l'intero frammento stuffer è stato sostituito con Dna esogeno (spi-), sono stati eliminati i geni red e gam. in teoria, non potrebbero propagarsi in RecBCD+ per due motivi, uno perchè manca gam che inibirebbe l'azione di RecBCD e l'altro perchè la mancanza di red non dà la possibilità a lambda spi- di effettuare la replicazione per ricombinazione omologa, che darebbe dimeri in grado di autoassemblarsi e permettere il packaging. in pratica invece lo possono fare, purchè il batterio RecBCD+ sia anche RecA+ e all'interno della sequenza del fago sia inserita questa regione ad alta frequenza di ricombinazione (hot spot). RecA e questo sito permettono il packaging e la formazione di nuove particella fagiche.
Spero di aver capito in modo giusto!!! Ti ringrazio molto per la disponibilità!!!   |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-18 MolecularLab.it |
 |
|
|









