RICERCA DI DOMINI FUNZIONALI
Se non si osserva un significativo grado di similarità con
una proteina di cui sia nota la funzione si possono cercare patterns
aminoacidici specifici di determinate famiglie di proteine o regioni
di similarità locale con domini noti.
Sono state create diverse banche dati che raccolgono tali elementi
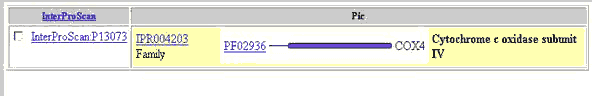
(PROSITE, PRODOM, PFAM,ecc.). Il sito della banca dati INTERPRO
raccoglie tutte le informazioni provenienti da tutte le banche dati
sopra citate.
- Predizione di Peptidi Segnale:
Per verificare la probabile presenza di un peptide segnale specifico
per l’indirizzamento della proteina al mitocondrio o ad
altri compartimenti cellulari possiamo utilizzare programmi specifici
come TARGETP
- Collegati al sito: http://www.cbs.dtu.dk/services/TargetP
- Ricerca di domini transmembrana:
Per ricercare domini transmembrana possiamo utilizzare diversi
programmi tra cui TMHMM
- Collegati al sito: http://www.cbs.dtu.dk/services/TMHMM
interpreta il risultato servendoti anche del link Help in alto
alla pagina
- Predizione della struttura secondaria:
Se non è nota la struttura terziaria della proteina in
esame o del suo omologo si possono ottenere informazioni rilevanti
sulla struttura e sulla funzione della proteina attraverso la
predizione della struttura secondaria.
Utilizzeremo il programma PSIPRED
- Collegati al sito: http://bioinf.cs.ucl.ac.uk/psiform.html
- Sottometti la sequenza proteica senza la riga di intestazione,
inserisci il tuo indirizzo e-mail e aspetta che ti arrivi il risultato
via posta.
- Collegati al sito: http://www.compbio.dundee.ac.uk/~www-jpred/submit.html
N.B.: per accedere al repertorio molto vasto di programmmi di
analisi per lo studio di proteine si può consultatre il
sito EXPASY ( http://www.expasy.org
) dove sono disponibili molti programmi per lo studio di proteine.
Un altro gruppo di programmi utili è disponibili presso
il CIRB di Bologna (http://gpcr.biocomp.unibo.it/predictors
).
- Consultazione di banche dati metaboliche:
Se vogliamo ottenere informazioni sulla funzione di una proteina
o sul complessomultienzimatico cui appartiene possiamo consultare
una banca dati metabolica come KEGG. - Collegati al sito http://www.genome.ad.jp/kegg/kegg2.html
- Naviga al suo interno alla ricerca di informazioni sulla funzione
di cox4
- Altre informazioni si possono ottenere consultando il database
LocusLink
dell NCBI.
- Nel caso di geni umani è possibile con GeneCard
individuare i prodotti genici, ed il loro causare malattie (utilissimo
per oncogeni). Mettendo insieme più banche dati fornisce
moltissime informazioni: dalla struttura 3D, alla loro espressione
nei vari tipi di tessuti, dagli omologi alle varianti SNPs.
ESERCIZI PROPOSTI:
·Determina funzione, struttura e localizzazione di questa proteina.
