-4° Giorno-
Ligazione del plasmide e trasformazione cellule E. coli
Questo esperimento ha come obbiettivo l'inserimento
di un frammento di DNA nel vettore di clonaggio pGEM7Zf. Il frammento
porta estremità generate dalla digestione con XbaI e quindi
verrà introdotto nel sito XbaI presente nella regione dei
Multiple Cloning Sites (Linker)
Protocollo:
Si preparerà una reazione di ligazione utilizzando il vettore
pGEM7Zf digerito con XbaI e defosforilato e un frammento di DNA
pttenuto da una digestione con il medesimo enzima. Per creare la
molecola di DNA ricombinante si userà la DNA
ligasi de fago T4. Alla fine della reazione si trasformeranno
cellule di Escherichia coli DH5alfa, sensibili all'antibiotico ampicillina.
A disposizione si hanno 3 soluzione di DNA:
- Soluzione1: DNA del vettore digerito con XbaI [concentrazione:
25ng/ul in tampone EB (Tris-HCl 10mM, pH8.5)]
- Soluzione2: DNA del vettore digerito con XbaI e defosforilato
con fosfatasi alcalina di gambero [concentrazione 25 ng/ul]
- Soluzione3: Frammento di DNA da clonare risospeso in tampone EB
alla concentrazione di 25ng/ul.
Prima di allestire la reazione di ligazione è ooportuno calcolare
la quantità di insert da utilizzare. Per avere un rapporto
molare tra vettore e inserto di 1:3, utilizzando 50 ng di vettore
e considerando che i DNA da inserire è lungo 1.8Kbp, si dovranno
utilizzare 90ng di inserto.
- Allestire le reazioni secondo lo schema:
| Reazioni |
DNA |
Tampone diluizione
(1X) |
Tampone ligazione |
Volume Finale |
Note |
| Vettore linearizzato |
2ul |
8ul |
10ul |
20ul |
Controllo dell'efficienza
di ligazione |
| Vettore linearizzato e defosforilato |
2ul |
8ul |
10 |
20ul |
Controllo della defosforilazione |
| Vettore defosforilato + frammento
(rapporto 1:£) |
2 ul + 3.5ul |
4.5ul |
10ul |
20ul |
Ligazione vettore-inserto |
- Miscelare su vortex per 2-3 secondi
- Aggiungere 1ul di T4 DNA ligasi e miscelare
- Centrifugare per 2 secondi in microcentrifuga
- Incubare a 25°C per 5minuti.
Protocollo della trasformazione
Questa procedura consente di introdurre DNA in cellule di E.coli.
Si testeranno le cellule competenti preparate il giorno precedente
utilizzando il plasmide circolare pGEM7Zf, e si useranno cellule
competenti già testate e conservate in un congelatore a -80°C
in presenza di glicerolo al 10% fino al momento dell'uso.
- Si preparano due provette Eppendorf da 1.5ml sterili e vi si
trasferisce 200ul dic ellule competenti preparate precedentemente.
- Aggiungere il DNA seguendo gli schemi riportati:
| Schema
trasformazione cellule preparate |
| Campioni |
DNA |
Note |
| No DNA |
- |
Controllo Negativo |
| Vettore |
10ng |
Controllo Positivo |
| Schema
trasformazione cellule testate |
| Campioni |
DNA |
Note |
| Vettore linearizzato |
Tutta la miscela |
Controllo Ligazione |
| Vettore linearizzatoe defosforilato |
Tutta la miscela |
Controllo defosforilazione |
Vettore defosforilato + frammento
(rapporto 1:3) |
Tutta la miscela |
Reazione di ligazione |
- Tenere le cellule in ghiaccio per 20 minuti.
- Sottoporle a schok termico a 42°C per 2 minuti.
- A temperatura ambiente aggiungere 800ul di terreno LB. In questo
modo si diluisce la concentrazione di cloruro di calcio permettendo
alle cellule di riprendersi ed esprimere i geni necessari alla
sucessiva selezione.
- Chiudere e miscelare delicatamente.
- Incubare a 37°C per 40-60 minuti.
- Usare 8 piastre dit erreno solido LB contenente Ampicillina
(100ug /ml), X-gal (substrato cromogeno della beta-galattosidasi)
e IPTG (induttore del promotore lac che regola l'espressione del'alfa-peptide):
- Con l'ausilio di un ansa sterile si distribuiscono 100 ul (equivalenti
a circa 1/10 del volume totale) omogeneamente, sulla superficie
del terreno. I rimanenti 9/10 vengono centrifigugati per 1 minuto
in microcentrifuga. Si elimina il surnatante e si rispospende
il pellet nel terreno rimasto. Le cellule vengono distribuite
con un'altra ansa sterile sulla piastra appositamente marcata.
- il controllo a cui non è stato aggiunto il DNA viene
centrifugato, il pellet risospeso nel terreno rimasto e piastrato
su un'unica piastra.. Allo stesso modo anche il controllo della
defosforilazione.
- Le piastre vengono poste per 12 ore a 37°C.
Note, risultati e conclusioni:
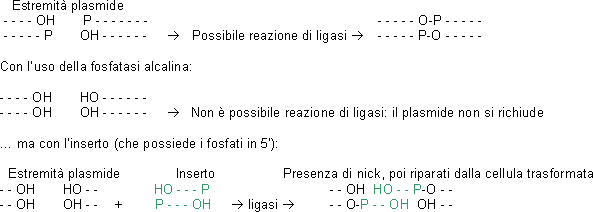
Nella ligazione che effettuiamo tra il plasmide pGEM 7Zf e un inserto
fornito, osserviamo anche l'attività della fosfatasi alcalina.
Questa, infatti, defosforilando gli OH presenti in 3' del plasmide,
linearizzato con XbaI non permette che questo dei richiudi su stesso.
Reazione di ligasi che avviene con la DNA ligasi del fago T4.
In condizioni di sterilità effettuiamo
poi la trasformazione delle cellule rese competenti il giorno
prima.
Usando il CaCl2 per rendere competenti le cellule di E. coli abbiamo
una efficienza di 106 trasformanti / µg di plasmide. Altre
tecniche come l'elettroporazione, permettono di avere efficienza
maggiori.
Per piastrare adottiamo questo schema, il quale ci permetterà
di osservare i risultati di ligazione e di contare più comodamente
le colonie:

