La replicazione del DNA avviene tramite un modello discontinuo e semiconservativo cioè nel prodotto derivato, la molecola figlia, vi è sempre una molecola di sintesi e una proveniente dallo stampo.
Questo modello venne dimostrato da M. Meselson e W.F. Stahl:
I due ricercatori hanno coltivato Escherichia Coli in azoto 14 e 15 (+ pesante), attraverso combinazioni e aspettando la replicazione hanno successivamente estratto il DNA e il risultato era che la sequenza di bande rispettava il modello semiconservativo, avendo densità H-L
Proteine che intervengono nella replicazione
DNA polimerasi in cellule procariote, ne conosciamo tre:
•polimerizzano i nucleosidi trifosfati (crea nuovi filamenti)
•lavorano, solo in direzione5’ 3’, ma devono incontrare un DNA a doppia elica (anche 3’ 5’)
La più conosciuta è la III questa, ha 10 subunità:
• alfa(attività polimerasica), teta (attività esonucleasica), sigma(funzione non nota); tutte e tre rappresentano il CORE polimerasi
• beta, aumenta la processività, cioè il numero dei nucleotidi aggiunti prima che si stacchi del filamento, della DNA polimerasi
• tao, tiene legati due DNA polimerasi (punti core e subunità beta)
•complesso gamma, composto da 5 subunità attacca e stacca la subunità beta nelle fasi della replicazione
Quando entrambi i filamenti saranno replicati completamente, le DNA polimerasi 3 subiranno un distacco delle subunità beta, ma il core rimarrà attaccato per una successiva replicazione
•DNA elicasi e proteine stabilizzanti dell’elica (A e B), favoriscono l’apertura dell’elica da copiare
•DNA ligasi, tramite l’ATP unisce frammenti di DNA
•DNA topoisomerasi, favorisce la risoluzione dei problemi derivanti dall’avvolgimento dell’elica, (SUPERAVVOLGIMENTI).
•RNA primasi, aggiunge inneschi di RNA (primer, oligonucleotidi), si attacca alle proteine stabilizzanti l’elica.
Meccanismi che intervengono nella replicazione
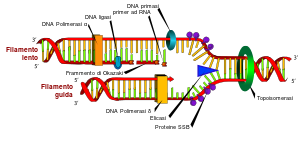

1) Svolgimento della doppia elica:
La proteine DNA A piglia contatto con la oriC, origine di replicazione, sulle sequenze ripetute 9 mer (basi) aprendo grazie all’ intervento di ATP la molecola di DNA nella 13 mer e costituendo così il complesso aperto; le regioni sono costituite di adenosina e timida (+ facili da scindere).
Sui due filamenti viene a posizionarsi di nuovo tramite una DNA C la elicasi (o DNA B), che svolge , idrolizzando l’ATP, il DNA procedendo in direzioni opposte dall’origine e formando il complesso di preavvio.
Le SSB (single strend binding, legano un filamento singolo), sono proteine che legano il DNA capaci di evitare il riappaiamento di due filamenti divisi attaccandosi su ognuno.
2)Replicazione nella forcella,: la forcella di replicazione ha forma di V con i suoi filamenti origine mentre quelli di nuova sintesi sono chiamati filamento guida (o veloce o leader), copiato grazie all’azione della DNA polimerasi dal filamento stampo (origine) 5’-3’ quindi avente direzione 3’-5’, e filamento in ritardo che siccome ha direzione opposta a quella in cui lavora la DNA polimerasi viene copiato attraverso un processo diverso ma che si ricongiunge a quello del filamento guida:
a.La RNA primasi aggiunge una serie di inneschi sul filamento in ritardo, lavorando in direzione 5’-3’
b.Si attaccano sull’estremità 3’ degli inneschi i frammenti di Okazaki
c.Gli inneschi sono rimossi dalla attività esonucleasica della DNA polimerasi 1, creando un GAP che poi viene riempito ,chiaramente partendo da 3’, dalla attività polimerizzante della stessa
d.Interviene la DNA ligasi che con l’uso di un a molecola di ATP realizza un legame fosfodiestere tra i frammenti di Okazaki
Se il GAP non fosse riempito, il filamento guida risulterebbe + corto del suo stampo di un lunghezza pari al suo innesco, e lo stesso problema si presenterebbe alla replicazione successiva. La molecola di DNA diventerebbe troppo corta per essere replicata e scomparirebbe dal genoma.
La lunghezza dei cromosomi è mantenuta costante da un blocco di sequenze ripetute in tandem che coprono le estremità, o telomeri di tutti gli enzimi eucariotici. Le telomerasi appunto aggiungono lr sequenze ripetu-te sul filamento stampo3’-5’.
Queste unità ripetute terminali sono tipicamente centinaia di copie di brevi sequenze ricche di basi azotate C e G, e si trovano nel filamento nucleotidico con l’estremità 3’ al telomero.


Aggiungo qualcosa…se posso,la replicazione del DNA avviene in maniera semiconservativa e discontinua..queste polimerasi sono batteriche,e differiscono da quelle eucariotiche in cui ce ne sono 5
Giusto,
la replicazione del DNA avviene in maniera semiconservativa e discontinua!
Grazie per la collaborazione!
Aggiornerò subito!
Ciao