11 December 2010 - 22:08

Non capita tutti i giorni che gli scienziati si mettano a parlare di alieni. Ma di “alieni” invece se ne è parlato molto negli ultimi giorni, in seguito ad un annuncio da parte della NASA, la ben nota agenzia spaziale americana. In realtà, la parola alieni è stata utilizzata solo a scopo divulgativo, probabilmente per destare l’attenzione del pubblico. Pur avendo a che fare con una scoperta di un certo interesse, non si tratta in realtà di alcun essere venuto dallo spazio. La notizia diffusa dagli scienziati americani riguarda piuttosto un batterio, più terrestre che mai, in grado di sopravvivere sostituendo il fosforo con l’arsenico per il suo sostentamento.
La scoperta rientra in quella disciplina nota come astrobiologia (o esobiologia), una scienza che ha come oggetto di studio la vita al di fuori del pianeta Terra. Come tutti noi sappiamo, al momento non esistono forme di vita extraterrestri conosciute, di conseguenza l’astrobiologia non si occupa direttamente della vita su altri pianeti, ma piuttosto lavora formulando teorie su dove e come la vita potrebbe esistere in altre parti dell’universo. Gli astrobiologi elaborano i dati che le osservazioni spaziali forniscono, dati sulla presenza di pianeti intorno ad altre stelle, sulla loro distanza dal proprio sole, e sulla composizione dell’atmosfera. Queste informazioni servono a far capire agli scienziati se altri pianeti possano o meno avere le caratteristiche geofisiche e chimiche che sulla Terra hanno permesso la nascita della vita, ipotizzando di conseguenza se la vita possa essere presente anche su quei corpi celesti. In questo senso l’astrobiologia potrebbe essere considerata una scienza fondamentalmente speculativa.
Le recenti scoperte della NASA sono state presentate, prima dell’annuncio, come qualcosa che avrebbe dato nuova linfa alla scienza dell’astrobiologia. Non si sapeva ancora in cosa consistesse tale scoperta, ma come è facile immaginare l’annuncio ha presto fatto il giro del mondo ed è stato accolto come una possibile rivelazione della scoperta di vita extraterrestre.
Così non è stato. Non è stata trovata nessuna forma di vita aliena su Marte, su Titano o su qualche altro corpo extraterrestre. Quello che avevano scoperto gli scienziati della NASA era piuttosto un batterio terrestre, vivente nel Mono Lake, un lago salato californiano. Piuttosto vicino, si direbbe, rispetto ai pianeti extrasolari. Anche se vivente sulla Terra, questo batterio è stato comunque chiamato con l’appellativo “alieno”. Questo non tanto per il luogo dove vive, ma piuttosto per le condizioni in cui è in grado di vivere. Il microrganismo in questione è un ceppo degli Halomonadaceae chiamato GFAJ-1. Esso è in grado di crescere anche quando la concentrazione di fosforo è troppo bassa per permettere la vita, usando per il suo sostentamento l’arsenico, che è presente in concentrazioni relativamente elevate nell’ambiente in cui vive.
Come sappiamo il fosforo, sotto forma di fosfato, è un elemento necessario per la vita. Esso fa parte della struttura fondamentale degli acidi nucleici (DNA e RNA) ed è determinante per il funzionamento di proteine e lipidi. L’ATP, la molecola usata da tutte le cellule viventi come unità di scambio dell’energia, funziona proprio usando il legame altamente energetico fra gruppi fosfati.
L’ambiente dove vive GFAJ-1 è un lago salato che contiene alte concentrazioni di arsenico (200 μM). Gli scienziati hanno inserito un inoculo di sedimenti lacustri in un terreno contenente glucosio, vitamine e tracce di metalli, ma completamente privo di fosfato e additivi organici. L’unico fosfato presente nel mezzo derivava da impurità dei suoi componenti salini, ed era in concentrazione di solo 3,1 μM.
Addizionando arseniato in concentrazioni diverse i ricercatori sono riusciti ad isolare il ceppo in questione, che è risultato in grado di crescere nel terreno contenente il fosforo in basse concentrazioni e addizionato con l’arsenico. Per verificare che l’arseniato venisse effettivamente acquisito dal microrganismo è stata usata la spettrometria di massa. L’utilizzo di arseniato marcato con un isotopo radioattivo di As, inoltre, ha permesso di osservare la localizzazione intracellulare dell’arsenico utilizzato dalle cellule. L’arsenico è stato osservato in frazionamenti cellulari di proteine, metaboliti, lipidi e acidi nucleici. La presenza dell’arsenico nella struttura del DNA è stata in seguito verificata con ulteriori metodi spettroscopici, mentre la concentrazione di fosforo nel DNA dei batteri cresciuti nel terreno quasi privo dell’elemento sono risultate incompatibili con la vita.
Bisogna sottolineare che, tuttavia, il batterio non è un arsenofilo obbligato. GFAJ-1 cresce più velocemente quando gli si fornisce fosforo invece di arsenico, anche se il fosforo è di un ordine di grandezza meno concentrato dell’arsenico. Questo perché il fosforo dà luogo a molecole più stabili di quelle contenenti arsenio, e quindi è un elemento migliore per la costruzione delle biomolecole. Tuttavia gli esperimenti condotti dalla NASA hanno dimostrato che il microrganismo scoperto è in grado di sostituire il fosfato con l’arseniato nelle sue molecole biologiche nel caso il fosforo scarseggi. L’arseniato deve dunque poter adempiere a tutte quelle funzioni chimiche e dare luogo alle stesse interazioni del normale fosfato all’interno delle cellule.
Quali conclusioni scientifiche, e forse anche filosofiche, potremmo trarre da queste osservazioni? La scoperta della NASA non è importante in quanto conferma diretta di vita aliena, come molti speravano prima che i dettagli della scoperta venissero diffusi. Essa è probabilmente importante su un livello concettuale. Questo infatti è forse l’indizio più consistente del fatto che la vita, forse, può funzionare anche in assenza di tutte quelle condizioni che sulla Terra sono fondamentali per gli organismi, come la presenza di una sufficiente concentrazione di fosforo. Questa scoperta apre la strada ad un modo diverso di lavorare e ad ipotesi più flessibili riguardo la presenza di vita nell’universo.
Il fosforo è stato da sempre considerato un elemento di fondamentale importanza per il DNA e l’RNA, le molecole alla base della vita. Nonostante sappiamo da tempo che alcuni elementi accessori alla vita, come il ferro, possono essere sostituiti con altri, fino a poco tempo fa pochi biologi, probabilmente, avrebbero preso in considerazione che molecole come DNA e RNA potessero funzionare anche senza fosforo. Per quanto ne sapevamo, infatti, la composizione di tali molecole fondamentali è la stessa in tutti gli organismi conosciuti.
Si tratta questa di “un’altra vita”? Dobbiamo considerare GFAJ-1 un organismo a parte rispetto a tutti gli altri? La risposta credo che sia negativa. Nonostante la strategia usata dal batterio per sopravvivere sia per il momento unica, dobbiamo ammettere che tale caratteristica deve essere stata acquisita secondariamente durante l’evoluzione. Il batterio condivide antenati comuni a tutti gli altri organismi terrestri, antenati che si sono evoluti, probabilmente, con un DNA di struttura tipica. Dunque gli alieni ancora non sono scesi sulla terra e GFAJ-1 è più terrestre che mai. Ma questa scoperta dopo tutto ci porta ad una riflessione: forse la vita ha più assi nella manica di quello che pensavamo. Forse in futuro ci riserverà altre inaspettate sorprese che, perché no, potrebbero riguardare ambienti esterni al nostro pianeta.
Tags:
Arsenico,
Astrobiologia,
DNA,
GFAJ-1,
NASA
24 October 2010 - 15:38
Dal 1953, anno in cui Watson e Crick pubblicavano la struttura tridimensionale della doppia elica del DNA, sono stati fatti ampi progressi nel campo della genetica. Questi passi avanti hanno portato, negli ultimi 60 anni, a nuove incredibili conoscenze, dalla definizione degli RNA catalitici negli anni ’80 fino al completamento del progetto genoma umano alcuni anni fa. Nonostante questi grandi progressi, e nonostante oggi sequenziare un genoma sia diventato quasi una routine in alcuni laboratori, il sequenziamento dei genomi ci ha insegnato un’importante lezione: la conoscenza anche completa della sequenza nucleotidica rappresenta solo il primo passo nella scoperta delle funzioni del DNA, molecola che sembra riservare sempre nuove sorprese agli scienziati che si occupano di genetica.
L’era postgenomica
La differenza di sequenza fra il genoma umano e quello delle scimmie è estremamente bassa. Questo porta in evidenza un importante interrogativo: cosa rende diverse specie così simili per la sequenza del DNA? Se in passato si credeva che sequenziando il genoma di un organismo si potesse comprenderne tutti i processi biologici, oggi sappiamo invece che il pattern di espressione proteico di due cellule con contenuto genomico simile può essere molto diverso e la sua diversità non può essere compresa a pieno osservando la sola sequenza del DNA. Questo è il motivo per il quale, da qualche anno, è nata una nuova importante disciplina, chiamata proteomica. La proteomica ha lo scopo di studiare il cosiddetto proteoma, vale a dire l’insieme delle proteine espresse nelle cellule in un determinato momento o nel contesto di determinati processi. Lo scopo è anche quello di caratterizzare i meccanismi di regolazione che portano, da uno stesso genoma, alla definizione di quadri di espressione proteica anche molto diversi in tessuti differenti o in momenti diversi dello sviluppo. Si pensi al bruco e alla farfalla: stesso organismo e stesso DNA, ma fenotipo decisamente diverso.

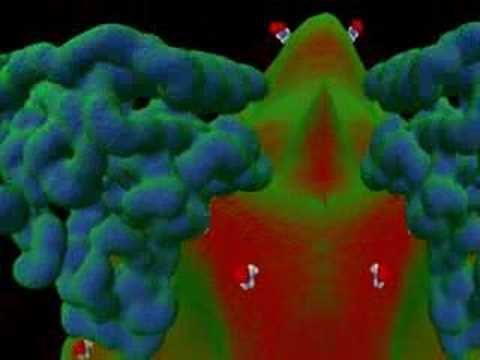
Modello tridimensionale di RISC, complesso proteico implicato nel silenziamento a microRNA
I nuovi livelli della regolazione genica
Tali meccanismi di regolazione dell’espressione genica sono in parte ancora da scoprire. Se infatti sappiamo già molto sulla regolazione della trascrizione basata sull’interazione dei fattori di trascrizione con specifiche sequenze a monte o a valle dei geni, c’è ancora molto da scoprire quando si prendono in considerazione altri livelli sui quali può agire la regolazione dell’espressione genica. Ad esempio, la sintesi di una proteina può essere regolata a livello traduzionale agendo sul blocco o sulla degradazione del suo mRNA. Una strategia di questo tipo fa uso dei cosiddetti RNA antisenso. Essi sono molecole polinucleotidiche in grado di legarsi a mRNA complementari, impedendone così la traduzione da parte dei ribosomi.
I microRNA sono un altro tipo di RNA regolatori. Essi sono in grado di riconoscere mRNA a sequenza complementare e impedirne la traduzione guidandoli verso la degradazione. Sebbene i microRNA siano conosciuti da tempo e siano usati anche a scopo sperimentale per il silenziamento genico, recentemente una nuova scoperta ha reso il quadro ancora più complesso. Alcuni scienziati del Medical Center di Boston, infatti, hanno scoperto che il ruolo di microRNA e mRNA può essere ribaltato: RNA trascritti, ma non codificanti, possono legarsi ai microRNA impedendo che essi possano operare il silenziamento genico, aggiungendo così una altro punto alla lista dei possibili meccanismi di regolazione.
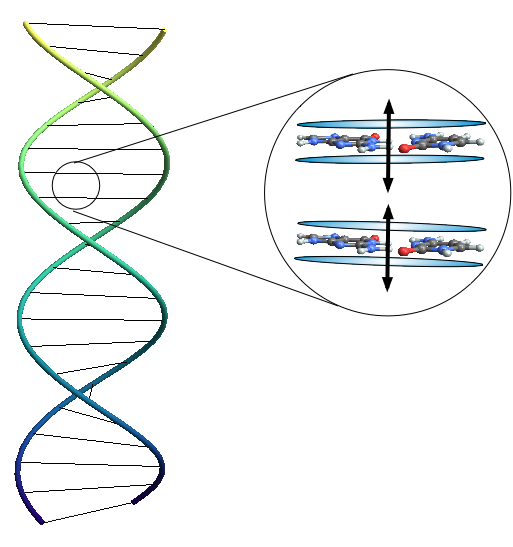
Applicando un modello di oscillatori armonici alla molecola del DNA, Elisabeth Rieper ne ha costruito un modello strutturale quantistico
La rivincita del DNA spazzatura
La scoperta di queste nuove molecole nucleotidiche implicate nella regolazione genica ha contribuito ad assegnare una funzione ad almeno una parte di quella porzione del genoma non codificante proteine, che negli anni passati era stata etichettata con il nome di junk DNA, vale a dire DNA spazzatura. Per molto si è discusso sulla possibile funzione di questa ampia porzione del genoma. Da molti era considerato nient’altro che DNA “egoista” che, simile ad un parassita, si era accumulato nei genomi pur non svolgendo alcuna funzione attiva, ma trasmettendosi di generazione in generazione insieme al DNA codificante. Altri ipotizzavano per il junk DNA una funzione di “tampone” per le mutazioni. Si era pensato cioè che il fatto che i geni fossero diluiti in mezzo ad una grande quantità di DNA inutile contribuisse ad abbassare la probabilità che una mutazione deleteria cadesse all’interno di un gene.
Oggi il DNA per il quale non si conosce una funzione sta diventando meno abbondante. Sempre più psuedogeni e sequenze di RNA non codificante vengono riconosciuti come responsabili della regolazione genica pur non sintetizzando proteine, ampliando così la porzione considerata funzionale del genoma.
Nuove frontiere: DNA quantistico
Una branca a dir poco avveniristica della biologia è la cosiddetta quantum biology, cioè “biologia quantistica”. Essa si prefigge di studiare l’implicazione dei fenomeni descritti dalla fisica quantistica all’interno dei sistemi biologici. Non accade spesso di sentir parlare di questo tipo di studi, ma i risultati ci sono e probabilmente ci aiuteranno in futuro a comprendere meglio i processi biochimici e la vita stessa.
Un fenomeno molto particolare del mondo dei quanti è il cosiddetto entanglement. Due particelle in stato di entanglement sono in grado, almeno apparentemente, di trasmettersi reciprocamente l’informazione sul proprio stato quantistico in maniera non-locale, cosa che da decenni assilla la mente dei fisici. Secondo uno studio condotto da Elisabeth Rieper della National University of Singapore il fenomeno dell’entanglement quantistico avrebbe un ruolo fondamentale nella strutturazione del DNA, affermando addirittura che secondo il modello strutturale classico la molecola del DNA non avrebbe energia sufficiente a rimanere unita.
Tutti questi nuovi dati e scoperte ci suggeriscono che il DNA, la molecola fondamentale della vita, racchiude ancora numerosi segreti che attendono di essere scoperti. Le osservazioni dei dati sperimentali, anche se ci forniscono nuove conoscenze, spesso introducono interrogativi nuovi, rendendo sempre più vario e complesso lo studio del fenomeno della vita.
Tags:
DNA,
entanglement,
junk DNA,
microRNA,
proteomica
20 February 2008 - 18:19
Gli eucarioti sono cellule che hanno una struttura interna complicata ed un nucleo definito delimitato dalla membrana, e si differenziano da procarioti che presentano una struttura semplice e un nucleo ben definito. Il DNA è sempre associato a proteine che negli eucarioti prende il nome di cromatina, che è costitituita da:
Dna, istoni e proteine non istoniche
Il DNA è il contenitore del materiale genetico, avvolto ad opera degli istoni. Gli istoni sono proteine cariche positivamente, quasi sempre, a pH basico e costituite da lisina arginina e un gruppo fosfato che reagisce con le cellule vicine. Esistono 5 tipi di istoni:
- H1, chiamato di giunzione
- H2A
- H2B
- H3
- H4
E in ognuno di questi vi è una regione polare (con carica positiva, a seconda dell’intensità) e una centrale non polare, che possono modificare i loro amminoacidi o rimanere costanti.
Il 2, 3, 4, 5 presi due volte ciascuno formano un ottamero, che è avvolto da 1 ¾ digiro di DNA, pari a 46 basi azotate. Gli ottameri sono uniti tra loro dal DNA link (o connette) composto da 15 a 55 bp (coppie di basi). L’ottamero più il DNA link viene chiamato nucleosoma, unità fondamentale della cromatina, che de un aspetto di collana di perle. H1 e DNA link uniscono i nucleosomi (il filo che unisce le perle della collana).
 Gli istoni agiscono direttamente sul DNA principalmente come elementi strutturali mantenendolo in precise forme, modificabili con ubiquitina, gruppi fosfati e sostanze biologicamente attive.
Gli istoni agiscono direttamente sul DNA principalmente come elementi strutturali mantenendolo in precise forme, modificabili con ubiquitina, gruppi fosfati e sostanze biologicamente attive.
Le proteine non istoniche differiscono principalmente dagli istoni in quanto hanno una carica prevalentemente negativa, formano l’impalcatura della cromatina e posseggono la forma di un cromosoma metafasico (la metafase una fase della mitosi,abbiamo i cromosomi completamente condensati e attaccati all’equatore del fuso mitotico).
Le proteine non istoniche si dividono in 3 gruppi:
- Proteine che regolano la trascrizione genica.
- Enzimi e fattori attivi nella trascrizione, replicazione, ricombinazione, riparazione del DNA e modificazione del DNA di prot. cromosomiche.
- Proteine che contribuiscono al mantenimento della struttura della cromatina o della conversione della cromatina dallo stato decondensato allo stato altamente compatto.
Ricordiamo che il dna può essere distinto in due forme:
- CIRCOLARE esistente nei procarioti, mitocondri, cloroplasti, virus (un cromosoma ad anello associato a proteine, una origine di replicazione).
- LINEARE lo troviamo nei virus, sia Single Strend che Double Strend, eucarioti (più origini di replicazione).
La cromatinaè una struttura altamente dinamica infatti il nucleosoma subisce delle modificazioni strutturali durante la trascrizione e la replicazione: un rilassamento che avviene in tre processi:
- Acetilazione, in generale degli istoni (lisina) che riduce la condensazione della cromatina caricandola positivamente ed eliminando la sua interazione con un gruppo fosfato del DNA (attrazione elettrostatica)
- Scissione di H1
- Rottura dei nucleosomi tramite i complessi enzimatici della trascrizione e replicazione che legano il DNA con una maggiore affinità delle proteine istoniche
La cromatina è modificata anche nel suo spessore e nella sua forma:
- Nella forma: il DNA trascritto diviene una fibra spessa e quello non trascritto una collana di perle
- Nello spessore: la cromatina condensata diventa di 30 mm, mentre la non condensata prende la forma di una collana di perle di 10 mm.
La cromatina viene anche distinta in due tipi a seconda del suo avvolgimento:
- Eucromatina, forma meno condensata rispetto alla seconda, contiene geni sempre attivi nella trascrizione
- Eterocromatina, divisa in:
- Facoltativa, dove i geni possono esprimersi
- Costitutiva, nella quale l’espressione genica non avviene
Quando viene estratta dalle cellule in soluzione tampone isotoniche, la cromatina appare condensata (30mm) e i suoi nucleosomi (allo stato nativo) sono impacchettati in una forma a spirale chiamata solenoide. Il solenoide presenta da
6 a 8 nucleosomi per giro e l’H1 legato al suo interno.
Tags:
cromatina,
DNA,
istoni,
packaging
15 February 2008 - 16:20
 Il meccanismo d’azione della Topoisomerasi II è veramente degno di nota. L’enzima prende due segmenti di DNA non contigui, rompe la doppia elica di uno di essi e trascina l’altro segmento nella breccia creata dal taglio; dopodichè l’enzima salda il taglio.
Il meccanismo d’azione della Topoisomerasi II è veramente degno di nota. L’enzima prende due segmenti di DNA non contigui, rompe la doppia elica di uno di essi e trascina l’altro segmento nella breccia creata dal taglio; dopodichè l’enzima salda il taglio.
In modo analogo la Topoisomerasi II può incatenare o disgiungere due molecole di DNA chiuse. Per svolgere il suo complicato lavoro, l’enzima utilizza energia dell’ ATP. Le topoisomerasi I, invece, non necessitano di ATP. Non possono stringere e sciogliere nodi di DNA a doppio filamento, ma possono comunque fare un lavoro simile con molecole circolari a singolo filamento. Fatto importantissimo, le Topoisomerasi di tipo I possono allentare la tensione torsionale del DNA a doppia elica. Questo tipo di tensione si instaura nella doppia elica in relazione al cambiamento dello stato topologico del DNA. La doppia elica sotto tensione da torsione dà luogo al fenomeno di superavvolgimento, che può essere compreso più facilmente nel caso di DNA circolare chiuso.
Tags:
DNA,
topoisomerasi
12 February 2008 - 18:35

Il DNA può annodarsi in diversi modi. Per esempio, una molecola di DNA a doppia elica pùo portare filamenti singoli, tra loro complementari, a entrambe le estremità 3′, cioè può avere terminazioni coesive. E’ il caso del DNA del batteriofago lambda quando il DNA è all’interno del batteriofago. Una molecola di quel tipo diventerà circolare spontaneamente in soluzione o all’interno del batterio quando il lambda DNA è iniettato nella cellula.
Durante il processo di trasformazione cacasuale del DNA lineare in un circolo chiuso, la molecola che ne risulta può mostrare di non essere annodata o di essere annodata in modi diversi.
La probabilità di annodarsi dipende dalla lunghezza del DNA e dal diametro effettivo del DNA. Il diametro effettivo del DNA può essere significativamente maggiore del suo diametro geometrico a causa della repulsione elettrostatica tra due segmenti di DNA in avvicinamento, ciascuno dei quali è fortemente carico negativamente. Questa repulsione elettrostatica è schermata da piccoli ioni positivi in soluzione o all’interno della cellula (Ioni Na). Tanto maggiore sarà la concentrazione di ioni Sodio, tanto più il diametro effettivo si avvicina al suo valore minimo geometrico 2 nm.
La probabilità di annodamento del DNA è stata calcolata accuratamente (Frank-Kamenetskii et al., 1975; Klenin et al., 1988) e questi calcoli sono stati pienamente confermati da misure sperimentali Della frazione di DNA annodato, in soluzioni a diverse concentrazioni di sodio (Rybenkov et al., 1993; Shaw e Wang, 1993). Poichè la trasformazione del DNA lineare in un circolo chiuso con estremità coesive avviene effettivamente in caso di infezione da parte di alcuni batteriofagi (non solo nel caso del fago lambda), la probabilità consistente, prevista teoricamente, di formazione di nodi, ha suggerito che deve esistere un particolare meccanismo in grado di intervenire nella topologia del DNA (Frank-Kamenetskii et al., 1975): Le DNA-Topoisomerasi di tipo II hanno dimostrato di essere enzimi in grado di stringere e di sciogliere i nodi del DNA (Liu et al., 1980; Wang, 1996).
Tags:
DNA,
forme-dna,
nodi,
topologia
10 February 2008 - 15:15
 Gli organismi attuali contengono molecole di DNA molto lunghe, costituite da milioni di coppie di basi (bp). Ne consegue che la replicazione di queste molecole giganti è associata a centinaia di migliaia di srotolamenti dei filamenti complementari gli uni sugli altri.
Gli organismi attuali contengono molecole di DNA molto lunghe, costituite da milioni di coppie di basi (bp). Ne consegue che la replicazione di queste molecole giganti è associata a centinaia di migliaia di srotolamenti dei filamenti complementari gli uni sugli altri.
Topoisomerasi I
Tale processo potrebbe portare inevitabilmente all’aggrovigliamento della molecola di DNA. Inoltre, in molti casi il DNA non ha affatto estremità libere: ciascuno dei filamenti complementari è chiuso in se stesso, con il risultato che i due filamenti di DNA sono legati tra di loro. Nel suo complesso, una molecola circolare chiusa di DNA può formare un nodo. La gravità dei problemi legati alla replicazione di lunghe molecole di DNA a doppia elica ha sorto dei dubbi sul fatto che il DNA all’interno della cellula assuma la configurazione a doppia elica di Watson & Crick.
La questione cruciale di come la cellula risolva i gravi problemi topologici legati a lunghe molecole lineari e a molecole circolari di DNA è stata risolta soprattutto attraverso lo studio della topologia di molecole di DNA-topoisomerasi, una nuova classe di enzimi (Wang, 1996) che modificano la topologia del DNA tagliando e risaldando i filamenti di DNA. Si distinguono in due classi di DNA-topoisomerasi: le topoisomerasi I tagliano e saldano uno dei due filamenti di DNA, quelle di tipo II tagliano e saldano entrambi i filamenti. E’ opinione generale che le DNA-topoisomerasi risolvano tutti i problemi topologici del DNA, annodamento e aggrovigliamento inclusi.
Tags:
DNA,
topoisomerasi
5 February 2008 - 14:53
Il DNA è il principio trasformante
Il DNA è stato scoperto nel 1953 dai dottori Watson e Crick, ma già nel 1928 esperimenti condotti dal dottor Griffith scoprirono che vi fosse l’esistenza di una molecola capace di trasformare geneticamente gli elementi;
Il principio trasformante
L’esperimento del dottor Griffith fu:
“aveva un batterio, lo pneumococco, del quale si distinguevano due ceppi, S che provocava polmonite quindi morte, R non virulento. Questi due tipi di ceppi vennero inoculati in un certo ordine provocando dei risultati.”

I risultati sono i seguenti:
- ceppo S -> topo morto;
- ceppo R -> topo vivo;
- ceppo S riscaldato (ucciso) -> topo vivo;
- ceppo S riscaldato con ceppo R -> topo morto.
L’ultimo risultato venne spiegato con un altro esperimento dal quale si dedusse che vi era una sostanza nel ceppo S riscaldato capace di modificare la struttura del DNA, e che faceva diventare il ceppo R nocivo.
Con questi esperimenti Griffith dimostrò che il materiale genetico era la sostanza del principio trasformante dei batteri. Erroneamente, come però la stragrande maggioranza degli scienziati suoi contemporanei, riteneva che questa sostanza dovesse essere di natura proteica.
La composizione del DNA
L’esperimento di Alfred D.Hershey e Martha Chase provò definitivamente nel 1953 che il materiale genetico è costituito da DNA e non da proteine. In seguito a questi risultati incontrovertibili anche gli scienziati che avevano criticato l’esperimento di Griffith si convincono dell’importantissimo ruolo biologico del DNA.
La sigla DNA significa acido desossiribonucleico ed è formato da polimeri (molteplici unità chimiche) di monomeri (singole unità chimiche) detti nucleotidi composti da:
- Gruppo fosfato
- Zucchero (pentoso a 5c)
- Base organica (basi azotate)
Il gruppo fosfato è legato allo zucchero da un legame fosfodiesterico (condensazione)
Nel DNA lo zucchero è il desossiribosio che in 2’ ha un gruppo enolico, mentre nell’RNA è il ribosio. Nello zucchero le posizioni 3’ 5’ sono i punti di legame con i nucleotidi precedenti e successivi, in posizione 1 è attaccata la base azotata.
Le basi azotate si distinguono in due classi:
- Purine, 2 anelli azoto-carbonio, l’azoto è sempre nella stessa posizione ( 9′ 3’ 1’ 7’), e sono:
- Adenina, in 6’ c’è un gruppo chetonico CO
- Guanina, in 6’ c’è un gruppo amminico NH2
- Pirimidine, 1 anello azoto-carbonio, lo zucchero si lega su 1, sono:
- Citosina, 5’ gruppo amminico e assenza di gruppo metile CH3 in 1’ o 5’
- Timina, 5’ gruppo chetonico
- Uracile, manca gruppo metile in 1’ o 5’
L’adenina, guanina e citosina le ritroviamo sia nel DNA sia nell’RNA, l’ uracile solo nell’RNA. Le basi azotate si strutturano in una doppia elica, due filamenti polinucleotidi associati che si avvolgono l’uno all’altro. Posizionate all’interno della struttura, all’ esterno c’è uno scheletro zucchero-fosfato.
Le basi azotate hanno delle regole (regole di Chagaff) precise per la loro strutturazione:
- Complementarietà, o adiacenza, adenina fronteggia timina e guanina la citosina perché vi sono problemi di dimensione in caso dovessimo invertire le posizioni; le basi infatti sono tenute insieme da legami a idrogeno differenti (2 per le prime, 3 per le seconde) che potrebbero modificare la stabilita della molecola di DNA
- Le purine sono più grandi delle pirimidine
- Contenuto di nucleotidi uguale nelle due categorie di basi azotate
Il nucleoside è una parte del nucleotide che comprende lo zucchero + la base azotata.
Entrambi hanno una nomenclatura appropriata: per esempio per i secondi si aggiunge il suffisso –ato .
Le forme del DNA
Esistono diverse isoforme del DNA in funzione della loro idratazione e situazione fisiologica/in-vitro in cui si trovano.

— Forma B:
destrosa ,10 basi per giro;
distanza base-asse 0,34 mm;
asse principale perpendicolare all’asse delle basi
filamenti antiparalleli (entrambe le direzioni 5’gr fosfato-3’gr ossidrilico)
solco minore e maggiore, nell’ultimo ci sono molecole per passaggi/contatti con altre cellule— Forma A: con scarsa umidità, sinistrosa, asse principale + inclinato, 11basi per giro
— Forma Z: sinistrosa, forma a zig zag per alternanza di purine e pirimdine
— Tripla elica: prodotta in laboratorio
La molecola del DNA è molto lunga, fino a qualche metro, e viene raggomitolata come un rocchetto da specifiche proteine.
La denaturazione del DNA, cioè lo svolgimento e la separazione dei filamenti che è chiamata anche fusione è un processo importante perché rende possibile la trascrizione attraverso il mRNA. La fusione è causata da un incremento di calore (in laboratorio) capace di rompere i legami idrogeno e le altre forze stabilizzanti il DNA, come legami tra basi adiacenti di uno stesso filamento.
La fusione modifica l’assorbanza della luce, misura direttamente proporzionale alla concentrazione del DNA.
Tags:
composizione-dna,
DNA,
forme-dna,
principio-trasformante






 Gli istoni agiscono direttamente sul
Gli istoni agiscono direttamente sul 
 Il meccanismo d’azione della
Il meccanismo d’azione della 
 Gli organismi attuali contengono molecole di
Gli organismi attuali contengono molecole di 
