10 April 2008 - 13:55
Uno dei delle domande esistenziali di una cellula è:
“Perchè una cellula dovrebbe suicidarsi?”
Non si tratta di masochismo cellulare, in certe circostanze la morte cellulare rappresenta un evento positivo per l’organismo.
Troviamo il fenomeno dell’apoptosi in un normale sviluppo embrionale, nella distruzione di cellule che rappresentano una minaccia per l’integritá dell’organismo; nel controllo della popolazione cellulare di un tessuto(omeostasi); nell’ eliminazione di cellule “Self” proprie “pericolose”
L’apoptosi è un sofisticato meccanismo in cui l’evoluzione cellulare ha fatto da setaccio per difendere l’organismo da cellule infettate da virus (ad opera di linfociti T citotossici); da cellule del sistema immune autoreattive; da cellule in cui si verifica un danno al DNA; da cellule tumorali.
L’Apoptosi è un processo attivo (richiede ATP) che viene indotto da segnali esogeni o endogeni e che serve ad eliminare, senza conseguenze cellule indesiderate. I processi fisiologici di regolazione dell’Apoptosi hanno segnali recettoriali, di danno direttamente al DNA e di segnali di membrana.
Se pensiamo allo sviluppo embrionale la morte cellulare è un processo fondamentale, per “scolpire” il nuovo organismo. Possiamo dire che la vita è un processo che in parte è reso possibile dalla morte. Infatti l’apoptosi durante lo sviluppo embrionale, per l’involuzione di strutture, può essere immaginata come una scultura di marmo; prima si parte dal blocco intero e poi piano piano si tolgono i pezzi con lo scalpello e il martello, così la scultura assume forma e sostanza. L’abbozzo delle mani è l’esempio più ecclatante per capire come funziona l’apoptosi nell’embriogenesi.
Altri segnali recettoriali di apoptosi sono nell’eliminazione dei neutrofilie macrofagi dopo l’infiammazione acuta, oppure nell’eliminazione dei linfociti T autoreattivi.
Per evitare l’accumolo di mutazioni che possono causare l’insorgenza di neoplasie, l’apoptosi gioca un ruolo fondamentale nell’eliminazione di cellule il cui DNA è stato danneggiato, cellule tumorali possono essere eliminate da parte dei linfociti T citotossici attraverso l’interazione di antigeni di membrana.
L’apoptosi é implementata mediante l’attivitá ordinata di
enzimi specifici, che agiscono prima della perdita dell’integritá della membrana cellulare.
Le caratteristiche morfologiche dell’apoptosi sono:
• Raggrinzimento cellulare;
• Condensazione e frammentazione della cromatina;
• Formazione di protuberanze (“blebs”) citoplasmatiche e di corpi apoptotici;
• Fagocitosi da parte di macrofagi o cellule non specializzate adiacenti
In vivo
– Le cellule “blebbing” sono ancora vitali (saggio del Trypan bleu);
– La transizione normale-raggrinzito-blebbing é rapida (10-30’), le cellule vengono poi fagocitate.
Istologicamente, l’apoptosi é quindi difficile da evidenziare a meno di un esteso coinvolgimento cellulare.
In vitro
Dopo il “blebbing”, c’é la vera e propria frammentazione cellulare in corpi apoptotici
• I corpi apoptotici vengono quindi fagocitati
• In situazioni -anche in vivo- in cui la apoptosi é massiva (es., nei tumori), cellule apoptotiche possono restare non fagocitate ed andare incontro a necrosi secondaria
In apoptosi c’è una progressiva degradazione proteica ad opera di proteasi specifiche (caspasi)
• Cross-linking proteico ad opera dellaTransglutaminasi II
• Frammentazione del DNA: formazione del “DNAladder”
• Attivazione di una “flippasi” e conseguente espressione di fosfatidilserina sulla parte esterna della membrana cellulare
Sperimentalmente si può evidenziare l’apoptosi di una determinata popolazione cellulare semplicemente osservando l’integrità del DNA
genomico per la presenza o assenza di attività endonucleasiche.

Perciò con un’ elettroforesi classica è possibile vedere i tagli delle endonucleasi mediate dalle caspasi che tagliano il DNA totale in frammenti di circa 200 paia di basi.
Line A DNA marker
Line B C Tipica strisciata di una cellula trattata con un apoptotico.
Linea D controllo Mock non trattato
Il metodo Tunel è una colorazione citochimica che sfrutta dei nucleotidi coniugati a digossigenina e aggiunti enzimaticamente al DNA ad opera della deoxynucleotidyl-transferase (TdT). Il DNA marcato viene rilevato grazie all’utilizzo di un anticorpo anti-digossigenina oppurtunamente coniugato con una molecola rivelatrice (fluorocromo, perossidasi ecc.ecc.)
L’apoptosi, è maggiormente resente in organismi cellulari, ma, in organismi monocellulari è presente in forme più o meno simili anche nei batteri, forse in difesa contro infezioni virali (a loro volta hanno escogitato numerosi stratagemmi per inattivare il programma apoptotico endocellulare)
Lo studio dell’apoptosi parte dal più famoso dei vermi trasparenti, il mitico Carnorabtis Elegans, durante lo sviluppo di C.Elegans, si formano 1090 cellule: 959resteranno a far parte del verme adulto e 131 vanno incontro ad apoptosi. Esperimenti di mutagenesi in cui il pattern di morte cellulare è alterato ha dato un grosso aiuto all’identificazione di regolazioni apoptotiche ritrovate anche nei mammiferi per analogia.

20 March 2008 - 22:17
La replicazione del DNA avviene tramite un modello discontinuo e semiconservativo cioè nel prodotto derivato, la molecola figlia, vi è sempre una molecola di sintesi e una proveniente dallo stampo.
Questo modello venne dimostrato da M. Meselson e W.F. Stahl:
I due ricercatori hanno coltivato Escherichia Coli in azoto 14 e 15 (+ pesante), attraverso combinazioni e aspettando la replicazione hanno successivamente estratto il DNA e il risultato era che la sequenza di bande rispettava il modello semiconservativo, avendo densità H-L
Proteine che intervengono nella replicazione
DNA polimerasi in cellule procariote, ne conosciamo tre:
•polimerizzano i nucleosidi trifosfati (crea nuovi filamenti)
•lavorano, solo in direzione5’ 3’, ma devono incontrare un DNA a doppia elica (anche 3’ 5’)
La più conosciuta è la III questa, ha 10 subunità:
• alfa(attività polimerasica), teta (attività esonucleasica), sigma(funzione non nota); tutte e tre rappresentano il CORE polimerasi
• beta, aumenta la processività, cioè il numero dei nucleotidi aggiunti prima che si stacchi del filamento, della DNA polimerasi
• tao, tiene legati due DNA polimerasi (punti core e subunità beta)
•complesso gamma, composto da 5 subunità attacca e stacca la subunità beta nelle fasi della replicazione
Quando entrambi i filamenti saranno replicati completamente, le DNA polimerasi 3 subiranno un distacco delle subunità beta, ma il core rimarrà attaccato per una successiva replicazione
•DNA elicasi e proteine stabilizzanti dell’elica (A e B), favoriscono l’apertura dell’elica da copiare
•DNA ligasi, tramite l’ATP unisce frammenti di DNA
•DNA topoisomerasi, favorisce la risoluzione dei problemi derivanti dall’avvolgimento dell’elica, (SUPERAVVOLGIMENTI).
•RNA primasi, aggiunge inneschi di RNA (primer, oligonucleotidi), si attacca alle proteine stabilizzanti l’elica.
Meccanismi che intervengono nella replicazione
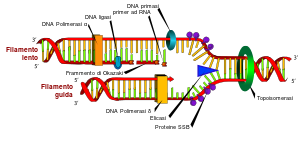

1) Svolgimento della doppia elica:
La proteine DNA A piglia contatto con la oriC, origine di replicazione, sulle sequenze ripetute 9 mer (basi) aprendo grazie all’ intervento di ATP la molecola di DNA nella 13 mer e costituendo così il complesso aperto; le regioni sono costituite di adenosina e timida (+ facili da scindere).
Sui due filamenti viene a posizionarsi di nuovo tramite una DNA C la elicasi (o DNA B), che svolge , idrolizzando l’ATP, il DNA procedendo in direzioni opposte dall’origine e formando il complesso di preavvio.
Le SSB (single strend binding, legano un filamento singolo), sono proteine che legano il DNA capaci di evitare il riappaiamento di due filamenti divisi attaccandosi su ognuno.
2)Replicazione nella forcella,: la forcella di replicazione ha forma di V con i suoi filamenti origine mentre quelli di nuova sintesi sono chiamati filamento guida (o veloce o leader), copiato grazie all’azione della DNA polimerasi dal filamento stampo (origine) 5’-3’ quindi avente direzione 3’-5’, e filamento in ritardo che siccome ha direzione opposta a quella in cui lavora la DNA polimerasi viene copiato attraverso un processo diverso ma che si ricongiunge a quello del filamento guida:
a.La RNA primasi aggiunge una serie di inneschi sul filamento in ritardo, lavorando in direzione 5’-3’
b.Si attaccano sull’estremità 3’ degli inneschi i frammenti di Okazaki
c.Gli inneschi sono rimossi dalla attività esonucleasica della DNA polimerasi 1, creando un GAP che poi viene riempito ,chiaramente partendo da 3’, dalla attività polimerizzante della stessa
d.Interviene la DNA ligasi che con l’uso di un a molecola di ATP realizza un legame fosfodiestere tra i frammenti di Okazaki
Se il GAP non fosse riempito, il filamento guida risulterebbe + corto del suo stampo di un lunghezza pari al suo innesco, e lo stesso problema si presenterebbe alla replicazione successiva. La molecola di DNA diventerebbe troppo corta per essere replicata e scomparirebbe dal genoma.
La lunghezza dei cromosomi è mantenuta costante da un blocco di sequenze ripetute in tandem che coprono le estremità, o telomeri di tutti gli enzimi eucariotici. Le telomerasi appunto aggiungono lr sequenze ripetu-te sul filamento stampo3’-5’.
Queste unità ripetute terminali sono tipicamente centinaia di copie di brevi sequenze ricche di basi azotate C e G, e si trovano nel filamento nucleotidico con l’estremità 3’ al telomero.
11 March 2008 - 01:18
Review

Per definizione è l’insieme di tutti i geni di un organismo e di tutte le sequenze intergeniche.
Il gene è quel tratto di DNA che codifica per la sintesi di un RNA o di una proteina
Nell’uomo ci sono 35-40000 geni che paragonati alla piccola negativa differenza di quantità dei nematodi (vermi) e dei pesci, e rapportando la complessità del nostro organismo con quello degli esempi ci si chiede quale sia il criterio che ci distingue.
La risposta è che la complessità del nostro organismo dipende dal prodotto (proteine) dei geni e non dal numero; infatti i geni si esprimono ognuno in maniera differente.
Si pensi anche che circa il 98% dei geni non si esprime, ma ha una funzione nella regolazione della stessa.
La quantità di DNA presente in un genoma (apolide) caratteristica di ogni specie è chiamata valore C , e la discrepanza tre grandezza gnomica e complessità genetica si chiama appunto paradosso C.
Il genoma è stato sequenziato nel 2001 da Collins e collaboratori, del NHRI (National Human Research In-stitute),scoprendo che il genoma è grande 3*10 alla nona bp (base pair = coppie di base, 3200 milioni di basi mb).
Il genoma sequenziato dell’uomo è di circa 2693 mb, e si divide in:
•25% GENICO
2,5% quello che si esprime
23% introni e pseudogeni
•75% INTRAGENICO
50% sequenze ripetute
24% duplicazione e altre sequenze
In definitiva il 97,5% dei geni del nostro corpo non si esprime, quindi rimane un 2,5% che sintetizza proteine. Comunque, quel 97,5% anche se non codifica il prodotto aiuta l’altra quota nell’espressione genica.
Ora, gli introni sono regioni non codificanti, gli esoni quelli codificanti, mentre gli pseudogeni rappresentano geni che non si esprimono (non sono attivi). Le sequenze ripetute sono principalmente divise in cinque classi:
a.Ripetizioni dovute a trasposoni (ripetizioni intersperse);
b.Ripetizioni di tipo (A)n, (CA)n, (CGG)n;
c.Duplicazioni (lunghezza 10-30000 kb)copiate da una regione all’altra del genoma;
d.Ripetizioni nei centromeri, estremità o intere braccia (cromosomi acrocentrici, cioè con braccia piccoline) chiamata RIPETIZIONE IN TANDEM;
e.Copie inattive di geni retrotrasposti, pseudogeni.
Analizzando la prima classe chiariamo che la trasposizione è un processo che permette lo spostamento del DNA mobile, costituente di una buona parte del genoma delle piante e degli animali superiori.
Il DNA mobile possiede due classi:
•Trasposoni
•Retrotrasposoni
Le differenze tra queste due classi risiedono nella differente modalità di inserimento della molecola di DNA:
il trasposone copia gli elementi direttamente in DNA,il retrotrasposone utilizza come intermedio una molecola di RNA cioè trascritte da una RNA polimerasi e converite di nuovo in DNA, per essere poi attaccate da nell’altra regione,da una trascrittasi inversa (questo processo, cioè il passaggio da una molecola di RNA ad una di DNA, nel 1975 non sembrava possibile).
RETROTRASPOSONI
•NON VIRALI
LINES
SINES (sequenze Alu)
•VIRALI
RIPETIZIONI LTR (Long Terminal Repeats)
I retrotrasposoni sono fonti di variabilità genetica, quindi anche di problemi in seguito a modificazioni non nella normalità.
Vengono distinti in:
Le ripetizioni LTR dei retroasposoni virali sono sequenze ripetute che permettono lo spostamento della molecola di DNA mobile, e rappresentano le porzioni terminali sia destra che sinistra della molecola. Al centro della stessa vi sono le proteine gag che hanno la funzione di codifica per la trascrittasi inversa.
Nei retroasposoni non virali, le “lines” sono sequenze ripetute di grandezza 6000-7000 bp (6-7 kb) e costi-tuiscono ben il 15% del genoma umano; le “sines”, 300-400 bp, sono principalmente rappresentate da se-quenze Alu cioè enzimi di restrizione che riconoscono la molecola di DNA e la tagliano.
LINE-Long Interdispersed Elements (6-7 kb)
SINE-Short Interdispersed Elements (300-400 bp)
20 February 2008 - 18:19
Gli eucarioti sono cellule che hanno una struttura interna complicata ed un nucleo definito delimitato dalla membrana, e si differenziano da procarioti che presentano una struttura semplice e un nucleo ben definito. Il DNA è sempre associato a proteine che negli eucarioti prende il nome di cromatina, che è costitituita da:
Dna, istoni e proteine non istoniche
Il DNA è il contenitore del materiale genetico, avvolto ad opera degli istoni. Gli istoni sono proteine cariche positivamente, quasi sempre, a pH basico e costituite da lisina arginina e un gruppo fosfato che reagisce con le cellule vicine. Esistono 5 tipi di istoni:
- H1, chiamato di giunzione
- H2A
- H2B
- H3
- H4
E in ognuno di questi vi è una regione polare (con carica positiva, a seconda dell’intensità) e una centrale non polare, che possono modificare i loro amminoacidi o rimanere costanti.
Il 2, 3, 4, 5 presi due volte ciascuno formano un ottamero, che è avvolto da 1 ¾ digiro di DNA, pari a 46 basi azotate. Gli ottameri sono uniti tra loro dal DNA link (o connette) composto da 15 a 55 bp (coppie di basi). L’ottamero più il DNA link viene chiamato nucleosoma, unità fondamentale della cromatina, che de un aspetto di collana di perle. H1 e DNA link uniscono i nucleosomi (il filo che unisce le perle della collana).
 Gli istoni agiscono direttamente sul DNA principalmente come elementi strutturali mantenendolo in precise forme, modificabili con ubiquitina, gruppi fosfati e sostanze biologicamente attive.
Gli istoni agiscono direttamente sul DNA principalmente come elementi strutturali mantenendolo in precise forme, modificabili con ubiquitina, gruppi fosfati e sostanze biologicamente attive.
Le proteine non istoniche differiscono principalmente dagli istoni in quanto hanno una carica prevalentemente negativa, formano l’impalcatura della cromatina e posseggono la forma di un cromosoma metafasico (la metafase una fase della mitosi,abbiamo i cromosomi completamente condensati e attaccati all’equatore del fuso mitotico).
Le proteine non istoniche si dividono in 3 gruppi:
- Proteine che regolano la trascrizione genica.
- Enzimi e fattori attivi nella trascrizione, replicazione, ricombinazione, riparazione del DNA e modificazione del DNA di prot. cromosomiche.
- Proteine che contribuiscono al mantenimento della struttura della cromatina o della conversione della cromatina dallo stato decondensato allo stato altamente compatto.
Ricordiamo che il dna può essere distinto in due forme:
- CIRCOLARE esistente nei procarioti, mitocondri, cloroplasti, virus (un cromosoma ad anello associato a proteine, una origine di replicazione).
- LINEARE lo troviamo nei virus, sia Single Strend che Double Strend, eucarioti (più origini di replicazione).
La cromatinaè una struttura altamente dinamica infatti il nucleosoma subisce delle modificazioni strutturali durante la trascrizione e la replicazione: un rilassamento che avviene in tre processi:
- Acetilazione, in generale degli istoni (lisina) che riduce la condensazione della cromatina caricandola positivamente ed eliminando la sua interazione con un gruppo fosfato del DNA (attrazione elettrostatica)
- Scissione di H1
- Rottura dei nucleosomi tramite i complessi enzimatici della trascrizione e replicazione che legano il DNA con una maggiore affinità delle proteine istoniche
La cromatina è modificata anche nel suo spessore e nella sua forma:
- Nella forma: il DNA trascritto diviene una fibra spessa e quello non trascritto una collana di perle
- Nello spessore: la cromatina condensata diventa di 30 mm, mentre la non condensata prende la forma di una collana di perle di 10 mm.
La cromatina viene anche distinta in due tipi a seconda del suo avvolgimento:
- Eucromatina, forma meno condensata rispetto alla seconda, contiene geni sempre attivi nella trascrizione
- Eterocromatina, divisa in:
- Facoltativa, dove i geni possono esprimersi
- Costitutiva, nella quale l’espressione genica non avviene
Quando viene estratta dalle cellule in soluzione tampone isotoniche, la cromatina appare condensata (30mm) e i suoi nucleosomi (allo stato nativo) sono impacchettati in una forma a spirale chiamata solenoide. Il solenoide presenta da
6 a 8 nucleosomi per giro e l’H1 legato al suo interno.
Tags:
cromatina,
DNA,
istoni,
packaging
15 February 2008 - 16:20
 Il meccanismo d’azione della Topoisomerasi II è veramente degno di nota. L’enzima prende due segmenti di DNA non contigui, rompe la doppia elica di uno di essi e trascina l’altro segmento nella breccia creata dal taglio; dopodichè l’enzima salda il taglio.
Il meccanismo d’azione della Topoisomerasi II è veramente degno di nota. L’enzima prende due segmenti di DNA non contigui, rompe la doppia elica di uno di essi e trascina l’altro segmento nella breccia creata dal taglio; dopodichè l’enzima salda il taglio.
In modo analogo la Topoisomerasi II può incatenare o disgiungere due molecole di DNA chiuse. Per svolgere il suo complicato lavoro, l’enzima utilizza energia dell’ ATP. Le topoisomerasi I, invece, non necessitano di ATP. Non possono stringere e sciogliere nodi di DNA a doppio filamento, ma possono comunque fare un lavoro simile con molecole circolari a singolo filamento. Fatto importantissimo, le Topoisomerasi di tipo I possono allentare la tensione torsionale del DNA a doppia elica. Questo tipo di tensione si instaura nella doppia elica in relazione al cambiamento dello stato topologico del DNA. La doppia elica sotto tensione da torsione dà luogo al fenomeno di superavvolgimento, che può essere compreso più facilmente nel caso di DNA circolare chiuso.
Tags:
DNA,
topoisomerasi
12 February 2008 - 18:35

Il DNA può annodarsi in diversi modi. Per esempio, una molecola di DNA a doppia elica pùo portare filamenti singoli, tra loro complementari, a entrambe le estremità 3′, cioè può avere terminazioni coesive. E’ il caso del DNA del batteriofago lambda quando il DNA è all’interno del batteriofago. Una molecola di quel tipo diventerà circolare spontaneamente in soluzione o all’interno del batterio quando il lambda DNA è iniettato nella cellula.
Durante il processo di trasformazione cacasuale del DNA lineare in un circolo chiuso, la molecola che ne risulta può mostrare di non essere annodata o di essere annodata in modi diversi.
La probabilità di annodarsi dipende dalla lunghezza del DNA e dal diametro effettivo del DNA. Il diametro effettivo del DNA può essere significativamente maggiore del suo diametro geometrico a causa della repulsione elettrostatica tra due segmenti di DNA in avvicinamento, ciascuno dei quali è fortemente carico negativamente. Questa repulsione elettrostatica è schermata da piccoli ioni positivi in soluzione o all’interno della cellula (Ioni Na). Tanto maggiore sarà la concentrazione di ioni Sodio, tanto più il diametro effettivo si avvicina al suo valore minimo geometrico 2 nm.
La probabilità di annodamento del DNA è stata calcolata accuratamente (Frank-Kamenetskii et al., 1975; Klenin et al., 1988) e questi calcoli sono stati pienamente confermati da misure sperimentali Della frazione di DNA annodato, in soluzioni a diverse concentrazioni di sodio (Rybenkov et al., 1993; Shaw e Wang, 1993). Poichè la trasformazione del DNA lineare in un circolo chiuso con estremità coesive avviene effettivamente in caso di infezione da parte di alcuni batteriofagi (non solo nel caso del fago lambda), la probabilità consistente, prevista teoricamente, di formazione di nodi, ha suggerito che deve esistere un particolare meccanismo in grado di intervenire nella topologia del DNA (Frank-Kamenetskii et al., 1975): Le DNA-Topoisomerasi di tipo II hanno dimostrato di essere enzimi in grado di stringere e di sciogliere i nodi del DNA (Liu et al., 1980; Wang, 1996).
Tags:
DNA,
forme-dna,
nodi,
topologia
10 February 2008 - 15:15
 Gli organismi attuali contengono molecole di DNA molto lunghe, costituite da milioni di coppie di basi (bp). Ne consegue che la replicazione di queste molecole giganti è associata a centinaia di migliaia di srotolamenti dei filamenti complementari gli uni sugli altri.
Gli organismi attuali contengono molecole di DNA molto lunghe, costituite da milioni di coppie di basi (bp). Ne consegue che la replicazione di queste molecole giganti è associata a centinaia di migliaia di srotolamenti dei filamenti complementari gli uni sugli altri.
Topoisomerasi I
Tale processo potrebbe portare inevitabilmente all’aggrovigliamento della molecola di DNA. Inoltre, in molti casi il DNA non ha affatto estremità libere: ciascuno dei filamenti complementari è chiuso in se stesso, con il risultato che i due filamenti di DNA sono legati tra di loro. Nel suo complesso, una molecola circolare chiusa di DNA può formare un nodo. La gravità dei problemi legati alla replicazione di lunghe molecole di DNA a doppia elica ha sorto dei dubbi sul fatto che il DNA all’interno della cellula assuma la configurazione a doppia elica di Watson & Crick.
La questione cruciale di come la cellula risolva i gravi problemi topologici legati a lunghe molecole lineari e a molecole circolari di DNA è stata risolta soprattutto attraverso lo studio della topologia di molecole di DNA-topoisomerasi, una nuova classe di enzimi (Wang, 1996) che modificano la topologia del DNA tagliando e risaldando i filamenti di DNA. Si distinguono in due classi di DNA-topoisomerasi: le topoisomerasi I tagliano e saldano uno dei due filamenti di DNA, quelle di tipo II tagliano e saldano entrambi i filamenti. E’ opinione generale che le DNA-topoisomerasi risolvano tutti i problemi topologici del DNA, annodamento e aggrovigliamento inclusi.
Tags:
DNA,
topoisomerasi
5 February 2008 - 14:53
Il DNA è il principio trasformante
Il DNA è stato scoperto nel 1953 dai dottori Watson e Crick, ma già nel 1928 esperimenti condotti dal dottor Griffith scoprirono che vi fosse l’esistenza di una molecola capace di trasformare geneticamente gli elementi;
Il principio trasformante
L’esperimento del dottor Griffith fu:
“aveva un batterio, lo pneumococco, del quale si distinguevano due ceppi, S che provocava polmonite quindi morte, R non virulento. Questi due tipi di ceppi vennero inoculati in un certo ordine provocando dei risultati.”

I risultati sono i seguenti:
- ceppo S -> topo morto;
- ceppo R -> topo vivo;
- ceppo S riscaldato (ucciso) -> topo vivo;
- ceppo S riscaldato con ceppo R -> topo morto.
L’ultimo risultato venne spiegato con un altro esperimento dal quale si dedusse che vi era una sostanza nel ceppo S riscaldato capace di modificare la struttura del DNA, e che faceva diventare il ceppo R nocivo.
Con questi esperimenti Griffith dimostrò che il materiale genetico era la sostanza del principio trasformante dei batteri. Erroneamente, come però la stragrande maggioranza degli scienziati suoi contemporanei, riteneva che questa sostanza dovesse essere di natura proteica.
La composizione del DNA
L’esperimento di Alfred D.Hershey e Martha Chase provò definitivamente nel 1953 che il materiale genetico è costituito da DNA e non da proteine. In seguito a questi risultati incontrovertibili anche gli scienziati che avevano criticato l’esperimento di Griffith si convincono dell’importantissimo ruolo biologico del DNA.
La sigla DNA significa acido desossiribonucleico ed è formato da polimeri (molteplici unità chimiche) di monomeri (singole unità chimiche) detti nucleotidi composti da:
- Gruppo fosfato
- Zucchero (pentoso a 5c)
- Base organica (basi azotate)
Il gruppo fosfato è legato allo zucchero da un legame fosfodiesterico (condensazione)
Nel DNA lo zucchero è il desossiribosio che in 2’ ha un gruppo enolico, mentre nell’RNA è il ribosio. Nello zucchero le posizioni 3’ 5’ sono i punti di legame con i nucleotidi precedenti e successivi, in posizione 1 è attaccata la base azotata.
Le basi azotate si distinguono in due classi:
- Purine, 2 anelli azoto-carbonio, l’azoto è sempre nella stessa posizione ( 9′ 3’ 1’ 7’), e sono:
- Adenina, in 6’ c’è un gruppo chetonico CO
- Guanina, in 6’ c’è un gruppo amminico NH2
- Pirimidine, 1 anello azoto-carbonio, lo zucchero si lega su 1, sono:
- Citosina, 5’ gruppo amminico e assenza di gruppo metile CH3 in 1’ o 5’
- Timina, 5’ gruppo chetonico
- Uracile, manca gruppo metile in 1’ o 5’
L’adenina, guanina e citosina le ritroviamo sia nel DNA sia nell’RNA, l’ uracile solo nell’RNA. Le basi azotate si strutturano in una doppia elica, due filamenti polinucleotidi associati che si avvolgono l’uno all’altro. Posizionate all’interno della struttura, all’ esterno c’è uno scheletro zucchero-fosfato.
Le basi azotate hanno delle regole (regole di Chagaff) precise per la loro strutturazione:
- Complementarietà, o adiacenza, adenina fronteggia timina e guanina la citosina perché vi sono problemi di dimensione in caso dovessimo invertire le posizioni; le basi infatti sono tenute insieme da legami a idrogeno differenti (2 per le prime, 3 per le seconde) che potrebbero modificare la stabilita della molecola di DNA
- Le purine sono più grandi delle pirimidine
- Contenuto di nucleotidi uguale nelle due categorie di basi azotate
Il nucleoside è una parte del nucleotide che comprende lo zucchero + la base azotata.
Entrambi hanno una nomenclatura appropriata: per esempio per i secondi si aggiunge il suffisso –ato .
Le forme del DNA
Esistono diverse isoforme del DNA in funzione della loro idratazione e situazione fisiologica/in-vitro in cui si trovano.

— Forma B:
destrosa ,10 basi per giro;
distanza base-asse 0,34 mm;
asse principale perpendicolare all’asse delle basi
filamenti antiparalleli (entrambe le direzioni 5’gr fosfato-3’gr ossidrilico)
solco minore e maggiore, nell’ultimo ci sono molecole per passaggi/contatti con altre cellule— Forma A: con scarsa umidità, sinistrosa, asse principale + inclinato, 11basi per giro
— Forma Z: sinistrosa, forma a zig zag per alternanza di purine e pirimdine
— Tripla elica: prodotta in laboratorio
La molecola del DNA è molto lunga, fino a qualche metro, e viene raggomitolata come un rocchetto da specifiche proteine.
La denaturazione del DNA, cioè lo svolgimento e la separazione dei filamenti che è chiamata anche fusione è un processo importante perché rende possibile la trascrizione attraverso il mRNA. La fusione è causata da un incremento di calore (in laboratorio) capace di rompere i legami idrogeno e le altre forze stabilizzanti il DNA, come legami tra basi adiacenti di uno stesso filamento.
La fusione modifica l’assorbanza della luce, misura direttamente proporzionale alla concentrazione del DNA.
Tags:
composizione-dna,
DNA,
forme-dna,
principio-trasformante
4 February 2008 - 14:02
 Cominciamo con aprire questo blog con le presentazioni.
Cominciamo con aprire questo blog con le presentazioni.
Mi chiamo Lorenzo, sono laureato in tecniche di laboratorio biomedico, ho deciso di continuare gli studi in Biologia Molecolare perché credo che sia il futuro per la comprensione dell’eziopatogenesi. Non sarò solo, nell’introdurvi nel viaggio della Biologia Molecolare e cellulare.
Scopo di questo blog è di dare una visione di insieme sullo studio delle molecole degli esseri viventi e le cellule stesse, a livello dei meccanismi molecolari alla base della loro fisiologia, concentrandosi in particolare sulle interazioni tra le macromolecole, ovvero proteine e acidi nucleici DNA e RNA. Scopo non ultimo è di dare utili informazioni riguardo una serie di tecniche che consentono la rilevazione, l’analisi, la manipolazione, l’amplificazione tramite Polimerase Chain Reaction e la copia o clonaggio degli acidi nucleici.
Le interrelazioni tra le discipline affini come la genetica e la biochimica non sono mai univoche, perché altri campi della biologia si intersecano con altre discipline biologiche: per esempio con la bioinformatica per l’elaborazione dell’enorme mole di dati che vengono prodotte con la fisica nello studio delle biomolecole a livello di struttura tridimensionale.
Perciò ci stiamo per addentrare in un viaggio tanto misterioso quanto affascinante. Ci sarà tempo per capire, discutere e scambiarci un po’ di opinioni su argomenti più o meno curiosi, intriganti o semplicemente informativi riguardo la ricerca nel campo della Biologia Molecolare.
Benvenuti in INSIDE THE CELL
Viaggio nella Biologia Molecolare e cellulare.
Tags:
blog,
inside-the-cell








 Gli
Gli 
 Il meccanismo d’azione della
Il meccanismo d’azione della 
 Gli organismi attuali contengono molecole di
Gli organismi attuali contengono molecole di 

 Cominciamo con aprire questo
Cominciamo con aprire questo