9 febbraio 2009 - 9:19 pm
Ciao a tutti! Oggi vi parlerò di un articolo che non tratta strettamente le neuroscienze, ma che propone una tecnica che sicuramente potrà trovare interessanti risvolti in questo campo. Questo post è la traduzione (abbastanza fedele) di quello che ho scritto originariamente (in inglese) sull’ottimo blog ReporterGene.
Il
calcium imaging è una tecnica che è decisamente maturata negli ultimi tempi, e di anno in anno vengono generati indicatori per il
calcio codificati geneticamente sempre più evoluti.
Uno dei vantaggi dell’utilizzo di indicatori codificati geneticamente è quello di poterli utilizzare per l’imaging in vivo e quindi per studiare processi fisiologici (o patologici) in un sistema molto più completo (e complesso) di -per esempio- una coltura cellulare o una fettina di tessuto.
Diversi approcci sono stati utilizzati per ottenere imaging in vivo, alcuni dei quali fanno uso di una fibra ottica, o “fibroscopio”, inserita nella regione da analizzare e collegata ad un microscopio. Questo è ovviamente un metodo invasivo, e limita l’osservazione alla piccola zona d’azione della fibra. Un altro approccio meno invasivo è quello di utilizzare obiettivi a lunga distanza focale per ottenere immagini di una regione del corpo esposta: come già visto, questo metodo è stato usato, ad esempio, per analizzare la plasticità delle spine dendritiche nella corteccia cerebrale. Anche questa tecnica, tuttavia, permette la visione di una limitata area di interesse, oltre a richiedere un intervento chirurgico piuttosto delicato.
L’approccio recentemente descritto da Rogers e colleghi su PlOS One, invece, è completamente non invasivo. Hanno infatti generato una linea di topi transgenici che esprimono in maniera selettiva (tamite un approccio floxed-stop) una proteina sensibile al calcio, detta GFP-aequorina. Il reporter può essere espresso selettivamente nelle cellule di interesse semplicemente incrociando questi topi con topi che esprimono Cre in quelle cellule (molte di queste linee sono disponibili commercialmente). La GFP-aequorina è una proteina che consiste di una parte sensibile al calcio (aequorina) che in presenza di ioni Ca2+  può ossidare un substrato (chiamato coelenterazina), producendo luminescenza, la quale eccita l’altra metà della proteina (GFP, green fluorescent protein) che quindi fluoresce.
può ossidare un substrato (chiamato coelenterazina), producendo luminescenza, la quale eccita l’altra metà della proteina (GFP, green fluorescent protein) che quindi fluoresce.
L’idea è quindi quella di iniettare coelenterazina e misurare la fluorescenza generata da GFP.
Questo metodo permette di osservare variazioni di calcio nell’intero animale, anche se ovviamente ha una risoluzione spazio-temporale più bassa (ma comunque buona) rispetto agli altri metodi sopra menzionati.
E in effetti sembra che funzioni bene! Quando GFP-aequorin è stata espressa nei mitocondri delle cellule muscolari, infatti, ha permesso di misurare l’aumento di calcio durante la contrazione muscolare. Questo è stato testato sia dopo una contrazione introdotta artificialmente, sia in condizioni fisiologiche come l’attività muscolare spontanea di topi neonati (vedi figura), sia in situazioni “patologiche”, come le convulsioni evocate da kainato.
Per meglio vedere questo sistema in azione consiglio fortemente di guardare i video allegati all’articolo (che come tutti gli articoli di PLoS è ad accesso gratuito).
Riassumendo, questo approccio permette di misurare il calcio in modo non invasivo e su tutto l’animale in condizioni fisiologiche e patologiche. Questa tecnica potrebbe trovare interessanti applicazioni in studi che misurano lo stato metabolico di un certo tessuto, o l’apoptosi o uno dei moltissimi processi regolati dal calcio (la memoria ad esempio). Il passo successivo, ovviamente, sarebbe quello di ottenere questo tipo di registrazioni in un sistema in cui l’animale si può muovere liberamente. Se avete visitato il link qui a sinistra avrete capito che non siamo lontani dall’ottenerlo, ed infatti lo stesso gruppo ha già pubblicato un articolo a riguardo (Roncali et al., J. Biomed. Opt. 2008)!
Tags:
Animali transgenici,
Calcio,
Imaging,
In vivo
11 aprile 2008 - 12:47 pm
Come abbiamo già detto in passati post i neuroni comunicano tra loro grazie alla generazione di “scariche elettriche”, chiamate potenziali d’azione. E’ quindi interessante avere la possibilità di generare artificialmente dei potenziali d’azione per studiare le proprietà di un certo neurone o di un network di neuroni. Ad esempio uno può stimolare il neurone 1 e vedere cosa succede al neurone 2 che vi è collegato.
Il classico approccio per fare ciò è utilizzare un elettrodo (un piccolo filamento di argento) inserito in una micropipetta di vetro dalla punta di pochi millesimi di millimetro di diametro e riempita di una soluzione conduttrice. Questo microelettrodo viene messo a contatto con la cellula (rompendone o meno la membrana a seconda del tipo di esperimento) e permette di iniettare con precisione corrente nella cellula, oltre a poterne rilevare l’attività elettrica spontanea.
Esistono però diversi altri approcci per eccitare o inibire un neurone che non richiedono l’utilizzo di un microelettrodo. I vantaggi di non usare microelettrodi sono molteplici, ma i principali sono: 1) andare a piazzare la minuscola punta dell’elettrodo sulla membrana cellulare non è proprio la cosa più semplice del mondo… 2) non si va a disturbare meccanicamente la cellula 3) sono più alla moda oggigiorno, tanto che qualcuno ne parla in un blog 
Uno di questi metodi è quello di utilizzare un neurotrasmettitore come, ad esempio, il glutamato. Possiamo “spruzzare” una piccola quantità di glutamato sulla cellula e questa, nella maggior parte dei casi, verrà eccitata. Esistono molte variazioni sul tema, ma oggi vi parlerò di un nuovo lavoro, apparso su Nature Methods di questo mese, che ha introdotto un nuovo interessante approccio.
Il lavoro in questione è il seguente: Photochemical control of endogenous ion channels and cellular excitability. – Fortin et al. – Nat. Methods 2008 Apr;5(4):331-8.
Gli autori hanno generato un “photoswitchable affinity label” (PAL), una piccola molecola che si lega selettivamente a canali sulla membrana del neurone che fanno passare gli ioni potassio (K+). In particolare, questa molecola lega un tipo di canali per il potassio (voltage-gated potassium channels) che sono sensibili a variazioni di voltaggio nella cellula. La normale funzione di questi canali è quella di riportare la cellula allo stato basale dopo la generazione di un potenziale d’azione. Insomma hanno un effetto “calmante” sull’attività cellulare.
PAL può legarsi selettivamente a questi canali e bloccarli, impedendo così il passaggio di potassio, risultando quindi in un eccitazione della cellula. La cosa interessante è che PAL è “photoswitchable”, cioè può essere “accesa” o “spenta” utilizzando luce di colori differenti. Questo è dovuto al fatto che PAL può cambiare forma quando viene colpita da luce di un certo colore: utilizzando luce viola PAL non blocca il canale, utilizzando luce verde lo blocca.
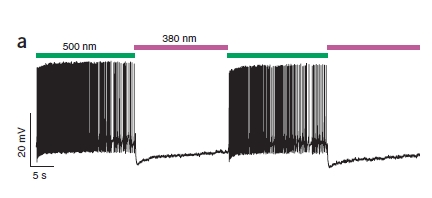
Il risultato è facilmente visualizzabile in questa figura:
Come vedete, quando la cellula è colpita da luce verde è iperattiva (ogni linea verticale rappresenta un potenziale d’azione, in alcune parti sono così fitti da non poterli distinguere) perchè PAL sta bloccando i canali al K+, mentre la luce viola silenzia la cellula.
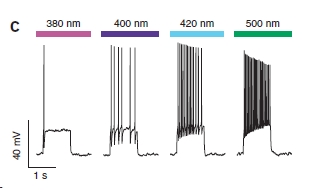
E non è finita così! Il funzionamento è graduale, quindi utilizzando luce di colore intermedio si ottiene una parziale eccitazione della cellula.
Certo, tutto questo è stato fatto su cellule in coltura ma sarebbe molto interessante vedere questo tipo di tecniche utilizzate in vivo (non dubito che ci si arriverà presto…). Ovviamente lo stesso principio potrebbe essere applicato a qualsiasi altro canale sui neuroni, si tratterà solo di sintetizzare molecole specifiche che permettano di legarsi ad altri canali!
Tags:
Attività neuronale,
Canali voltaggio dipendenti,
Elettrofisiologia,
Firing,
Imaging,
Neuroscienze,
Potenziali d'azione
2 febbraio 2008 - 11:44 pm
 Negli ultimi 20 anni la biologia molecolare ha fatto passi da gigante. Siamo arrivati ad un punto in cui parlare di DNA è cosa normale, tanto che uno si può far sequenziare il genoma ad un prezzo relativamente abbordabile, ci si può costruire il proprio batterio personale e chiunque ha la propria opinione sugli OGM (anche se magari non sa cosa siano…).
Negli ultimi 20 anni la biologia molecolare ha fatto passi da gigante. Siamo arrivati ad un punto in cui parlare di DNA è cosa normale, tanto che uno si può far sequenziare il genoma ad un prezzo relativamente abbordabile, ci si può costruire il proprio batterio personale e chiunque ha la propria opinione sugli OGM (anche se magari non sa cosa siano…).
Anche le neuroscienze sono state colpite da questa ondata di novità, tanto che oramai è cosa assolutamente normale e quasi necessaria utilizzare animali transgenici per molti tipi di esperimenti.
C’è però un’area delle neuroscienze che forse non ha ricevuto questo grande successo di pubblico ed è l’imaging. L’imaging è un campo molto vasto, che non è solo ristretto alle neuroscienze e che comprende una serie di tecniche atte a visualizzare molti processi cellulari. Pur non essendo necessariamente ristretto alle neuroscienze, credo che negli ultimi anni molti dei maggiori progressi a riguardo siano stati fatti proprio in questo campo.
Penso che uno dei motivi per cui queste tecniche siano un po’ sconosciute a molti (o solo conosciute di nome) sia la difficoltà tecnica dei processi coinvolti. Molte di queste tecniche richiedono l’uso di un microscopio ma, mentre ci sono poche difficoltà a capire come funziona un microscopio tradizionale e magari uno a fluorescenza, quando si comincia a parlare di eccitazione a due fotoni, deflettori opto-acustici e amenità del genere generalmente si perde molto velocemente l’attenzione di chi non è del campo. Se è vero infatti che fare un esperimento di imaging non richiede generalmente particolari conoscenze tecniche, per riuscire ad ottimizzare ed usare al meglio queste tecniche è necessaria una conoscenza piuttosto ampia dei processi fisici sottostanti. Ebbene sì, la fisica quantistica è molto importante in questo caso ed è cosa piuttosto normale oramai trovare articoli di microscopia applicata alle neuroscienze che sembrano trattare di elettronica o fisica più che di biologia! Personalmente sono affascinato da tutte queste tecniche, e credo che nei prossimi anni con nuovi avanzamenti tecnologici si riusciranno a fare cose ancora più incredibili di quanto si riesca a fare ora.
Tanto per farvi un paio di esempi, è oggigiorno facilmente possibile usare l’imaging per misurare il livello di ioni (come calcio, cloro, magnesio e zinco) all’interno di una cellula, all’interno di singoli compartimenti della cellula o addirittura visualizzare l’apertura di un singolo canale ionico. E’ possibile “visualizzare” l’attività elettrica di un neurone senza usare elettrodi, usando particolari coloranti sensibili al voltaggio. E’ possibile visualizzare singole molecole e vedere come si muovono all’interno di una cellula. E’ inoltre possibile visualizzare la crescita e plasticità dei neuroni al passare del tempo, come dicevo in precedenza… e potrei andare avanti per pagine e pagine!
Tutte queste cose sono relativamente facili su sistemi in vitro (es. cellule in coltura), tuttavia è ora possibile applicarne alcune su animali anestetizzati o addirittura svegli, ed è quindi possibile osservare questi processi cellulari in funzione durante determinate attività.
Insomma le possibilità sono tantissime e veramente affascinanti, e credo che nei prossimi anni ci sarà veramente un boom di nuove tecniche in questo campo… dobbiamo solo aspettare e vedere!
Tags:
Animali transgenici,
Biologia molecolare,
Imaging,
Neuroscienze
 Commenti disabilitati
Commenti disabilitati
18 gennaio 2008 - 8:52 am
E’ decisamente corretto dire che le cellule del nostro organismo sono sempre in continua attività e questo è ancora più vero nel caso dei neuroni del nostro cervello. L’attività del il cervello è determinata dai collegamenti fra i vari neuroni, che avvengono alle sinapsi. La maggior parte dei neuroni possiede dei processi molto ramificati, dette dendriti, che partono dal corpo cellulare e che funzionano come “sensori” per input da parte di altri neuroni. La superficie dei dendriti non è liscia, bensì contiene delle piccole estroflessioni di varia forma (spesso “a fungo”) chiamate spine dendritiche; è proprio su queste spine che vengono formate molte delle sinapsi eccitatorie con altri neuroni. Le spine contengono quella che viene chiamata post-synaptic density o PSD un complesso di molte proteine che sono coinvolte nei processi di formazione e rimodellamento delle sinapsi molto importanti -come si diceva in post precedenti- nei processi quali la memoria e l’apprendimento. Una delle più importanti fra queste proteine è PSD-95, ed è proprio questa proteina l’oggetto dello studio di cui vi parlerò oggi.
Lo studio è stato svolto nel laboratorio di Karel Svoboda, una delle figure più di spicco nel mondo dell’imaging neuronale. Potete trovare il lavoro completo al seguente link:
Rapid Redistribution of Synaptic PSD-95 in the Neocortex In Vivo – Gray et al. – PLoS Biology, 2006 Nov;4(11):e370
Gli autori iniziano con il generare dei topi transgenici che esprimono delle proteine fluorescenti nei neuroni di una parte della corteccia cerebrale deputata al processamento di stimoli sensoriali che nel topo sono mediati dalla stimolazione delle vibrisse. In particolare inseriscono una proteina fluorescente rossa chiamata mCherry che si distribuisce in tutta la cellula e serve per permettere di visualizzare i neuroni, e PSD-95-GFP, una PSD-95 fusa con una proteina fluorescente verde chiamata GFP. In questo modo è possibile visualizzare i dendriti dei neuroni, che saranno rossi, e la presenza di PSD-95 solo nelle spine. La tecnica usata è molto sofisticata in quanto grazie all’utilizzo di un microscopio a due fotoni permette di vedere queste proteine in vivo e di guardare la stessa cellula al passare dei giorni.

La prima cosa che hanno osservato è che, durante il periodo osservato (topi giovani) c’è una grande plasticità neuronale. La figura qui a sinistra mostra un dendrita in rosso con diverse spine, identificate dai punti verdi, corrispondenti alla presenza di PSD-95. Lo stesso dendrita è stato fotografato più volte a diversi giorni di vita dell’animale (le sigle P13, P15 etc. indicano un animale di 13 giorni, 15 giorni e così via). Nella colonna C si vede come al passare dei giorni la localizzazione di alcune spine cambi radicalmente, e questo è indice di un rimodellamento delle connessioni fra i neuroni o, per usare un termine che piace tanto a chi studia neuroscienze, di plasticità neuronale.
Al contrario, altre spine dello stesso dendrita (colonna D) sono più stabili, anche se possono cambiare di intensità e forma di giorno in giorno.
Avendo visto che il sistema funzionava e gli permetteva di vedere questi fenomeni di plasticità, hanno deciso di utilizzarlo per risolvere una questione nota da tempo: è noto infatti che le sinapsi possono apparire e sparire, alcune durano meno di un giorno, altre possono esistere per mesi (forse anni), ma le proteine della PSD, tuttavia, stanno nella sinapsi solo poche ore. Ciò non è dovuto ad una loro degradazione, che avviene dopo giorni, bensì come dimostrato in questo studio, ad una rapida diffusione al di fuori della spina e al passaggio nelle spine adiacenti. Per dimostrare ciò gli autori usano una tecnica molto interessante, chiamata fotoattivazione. In pratica usano un’altra variante di PSD-95, questa volta coniugata a paGFP, una proteina normalmente non fluorescente, ma che può essere attivata dalla luce, diventando fluorescente. E’ quindi possibile andare a colpire con un laser solo una spina, attivare la paGFP e misurarne la diffusione al di fuori della spina.

Come vedete nella parte sinistra dell’immagine qui a fianco, è possibile attivare selettivamente una spina (nell’esempio sono attivate 2 spine) e mostrare che la fluorescenza diminuisce nel tempo. Ma questo non è tutto! Come è più evidente nella parte destra dell’immagine, la fluorescenza si può spostare da una spina all’altra! Le spine più grandi tendono a trattenere di più PDS-95 rispetto a quelle più piccole, e sono quindi più stabili. Inoltre il processo non è semplicemente una pura diffusione di molecole, ma è reso più complesso dall’interazione con altre proteine presenti nella PSD.
Infine, l’ultima cosa mostrata nell’articolo è che questi processi sono dipendenti dall’esperienza e da stimoli esterni. Il tempo di ritenzione della proteina all’interno delle spine aumenta infatti con l’età, quando quindi le sinapsi diventano più stabili, ma diminuisce in topi adulti a cui sono state tagliate le vibrisse e che quindi sono in una situazione di deprivazione sensoriale.
Devo dire che, nonostante sia anche io nel campo dell’imaging, e quindi un po’ di parte, ritengo che poter vedere questi fenomeni con i propri occhi sia semplicemente una cosa formidabile.Uno dei punti forti di questo lavoro, a mio parere, sta nel fatto che sono riusciti a fare tutto ciò in vivo e mostrare quindi che questi fenomeni di diffusione avvengono anche nella situazione reale e non sono solo un “artefatto” dei sistemi di colture cellulari (cosa che accade più spesso di quanto si voglia ammettere).
Tags:
GFP,
Imaging,
Neuroscienze,
Plasticità sinaptica
20 novembre 2007 - 9:49 pm
Immaginate di voler capire come funziona un apparecchio elettronico:
lo aprite e ciò che vedete è un ammasso di cavi che vanno da ogni parte… una cosa che potreste fare per cominciare è disegnare una mappa dei fili, segnandovi da dove partono e dove arrivano. Certo, non sarebbe la soluzione finale al vostro problema, ma potrebbe essere un buon punto di partenza.
Studiando il cervello ci si trova spesso di fronte ad un problema simile, ma molto più complesso: un cervello umano contiene approssimativamente 100 miliardi di neuroni di molti diversi tipi e si stima che ciascuno di essi abbia collegamenti (sinapsi) in media con altri 7000 neuroni. Una delle sfide più grandi è quindi capire come questi neuroni siano collegati tra di loro, in modo da poter creare una mappa delle connessioni del cervello.
Un enorme passo avanti in questo senso è stato annunciato in un recente studio apparso sull’ultimo numero di Nature da parte di Jean Livet e colleghi al Dipartimento di Biologia Molecolare e Cellulare dell’Università di Harvard (e con uno studio del genere non c’è da sorprendersi che abbiano avuto l’immagine di copertina…).
 L’articolo riporta la generazione di varie linee di topi transgenici chiamati “Brainbow”, nome derivato dall’unione di brain (=cervello) e rainbow (=arcobaleno). I neuroni dei topi Brainbow, infatti, emettono fluorescenza in 100-150 colori differenti! Ciò è possibile grazie all’espressione nei neuroni di questi topi di diverse proteine fluorescenti: GFP (verde), YFP (gialla), CFP (azzurro), OFP (arancio) e RFP (rossa). Queste proteine vengono fatte esprimere in modo casuale ed a diversi livelli nei diversi neuroni per generare i diversi colori.
L’articolo riporta la generazione di varie linee di topi transgenici chiamati “Brainbow”, nome derivato dall’unione di brain (=cervello) e rainbow (=arcobaleno). I neuroni dei topi Brainbow, infatti, emettono fluorescenza in 100-150 colori differenti! Ciò è possibile grazie all’espressione nei neuroni di questi topi di diverse proteine fluorescenti: GFP (verde), YFP (gialla), CFP (azzurro), OFP (arancio) e RFP (rossa). Queste proteine vengono fatte esprimere in modo casuale ed a diversi livelli nei diversi neuroni per generare i diversi colori.
Topi ed altri animali le cui cellule esprimono proteine fluorescenti non sono certo una novità, negli ultimi 10-15 anni si è visto un aumento esponenziale del loro numero. La novità di questo studio è il fatto di avere un così grande numero di colori diversi che permette di seguire facilmente il percorso di un neurone, capire da dove parte e dove arriva e con quali altri neuroni viene a contatto. Sarà infatti probabile che neuroni vicini tra di loro abbiano diverso colore, rendendo così più “semplice” la mappatura delle varie connessioni.
L’articolo mostra anche esempi pratici d’uso di Brainbow come la ricostruzione in 3D di alcuni network cellulari nel cervelletto ed uno studio dell’interazione neuroni-glia.
Per chi fosse interessato all’aspetto più tecnico: il trucco sta nell’utilizzo di un transgene contenente le diverse XFP messe in sequenza in modo tale che solo la prima proteina possa essere tradotta. Le varie XFP sono messe all’interno di siti lox incompatibili tra loro (loxP, lox2272 e loxN) e mutualmente esclusivi; questo in pratica vuol dire che se Cre excide il DNA all’interno di una coppia di questi siti, gli altri non sono più funzionali. Il taglio può avvenire con la stessa probabilità a ciascun sito, portando così all’espressione di una diversa XFP in neuroni diversi. Aggiungete il fatto che diverse copie del transgene possono essere inserite in tandem (una delle linee riportate nell’articolo ha 8 copie del transgene) ed eccovi tutta la variabilità di espressione necessaria per ottenere 100 o più colori diversi!
Una domanda mi sorge però spontanea: supponendo di arrivare ad avere una mappa completa delle connessioni cerebrali, non credete ci troveremo poi in una situazione di stallo su come analizzare la inimmaginabilmente immensa quantità di dati che deriverebbe da questo studio? E come memorizzare questa enorme quantità di dati in un modo accessibile e funzionale?
Ad ogni modo, per ora la cosa migliore da fare è godersi un paio delle fantastiche immagini dei neuroni Brainbow!
E per chi volesse, ecco il link all’articolo (è necessaria una subscription a Nature per vedere il full-text):
Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system – Nature, 2007

Tags:
Animali transgenici,
Biologia molecolare,
Imaging,
Neuroscienze
 può ossidare un substrato (chiamato coelenterazina), producendo luminescenza, la quale eccita l’altra metà della proteina (GFP, green fluorescent protein) che quindi fluoresce.
può ossidare un substrato (chiamato coelenterazina), producendo luminescenza, la quale eccita l’altra metà della proteina (GFP, green fluorescent protein) che quindi fluoresce.




 Negli ultimi 20 anni la
Negli ultimi 20 anni la 

 L’articolo riporta la generazione di varie linee di topi transgenici chiamati “Brainbow”, nome derivato dall’unione di brain (=cervello) e rainbow (=arcobaleno). I neuroni dei topi Brainbow, infatti, emettono fluorescenza in 100-150 colori differenti! Ciò è possibile grazie all’espressione nei neuroni di questi topi di diverse proteine fluorescenti: GFP (verde), YFP (gialla), CFP (azzurro), OFP (arancio) e RFP (rossa). Queste proteine vengono fatte esprimere in modo casuale ed a diversi livelli nei diversi neuroni per generare i diversi colori.
L’articolo riporta la generazione di varie linee di topi transgenici chiamati “Brainbow”, nome derivato dall’unione di brain (=cervello) e rainbow (=arcobaleno). I neuroni dei topi Brainbow, infatti, emettono fluorescenza in 100-150 colori differenti! Ciò è possibile grazie all’espressione nei neuroni di questi topi di diverse proteine fluorescenti: GFP (verde), YFP (gialla), CFP (azzurro), OFP (arancio) e RFP (rossa). Queste proteine vengono fatte esprimere in modo casuale ed a diversi livelli nei diversi neuroni per generare i diversi colori.