I neuroni umani spaccano. Controllate voi stessi! (ovvero: non bisogna essere geni per manipolare dei geni)
Misterioso ed affascinante il mestiere del ricercatore! Anche seguire gli avanzamenti della ricerca sui siti di divulgazione fa luccicare gli occhi e battere forte il cuore (almeno a quelli che non sono impegnati a rubare cani). A volte però i titoli originali citati dai lavori divulgativi spaventano e respingono, privandoci del guizzo di mettere anche noi il ditino dove l’hanno messo i ricercatori.
Ho trovato questa succosa notizia [1]: “La duplicazione parziale di un gene, avvenuta circa 2,5 milioni di anni fa, sarebbe responsabile dello sviluppo di alcune funzioni critiche che hanno permesso al cervello umano un salto di qualità rispetto a quello dei nostri più prossimi parenti.”
La notizia è tutta qua, per quanto ci si possa ricamare sopra: oggi ne sezioneremo brevemente il titolo e faremo qualche verifica per nostro conto. Insieme.
Il cervello: la causa di, e la soluzione a tutti i nostri problemi. Fonte foto: http://tumoricanegatto.blogspot.it
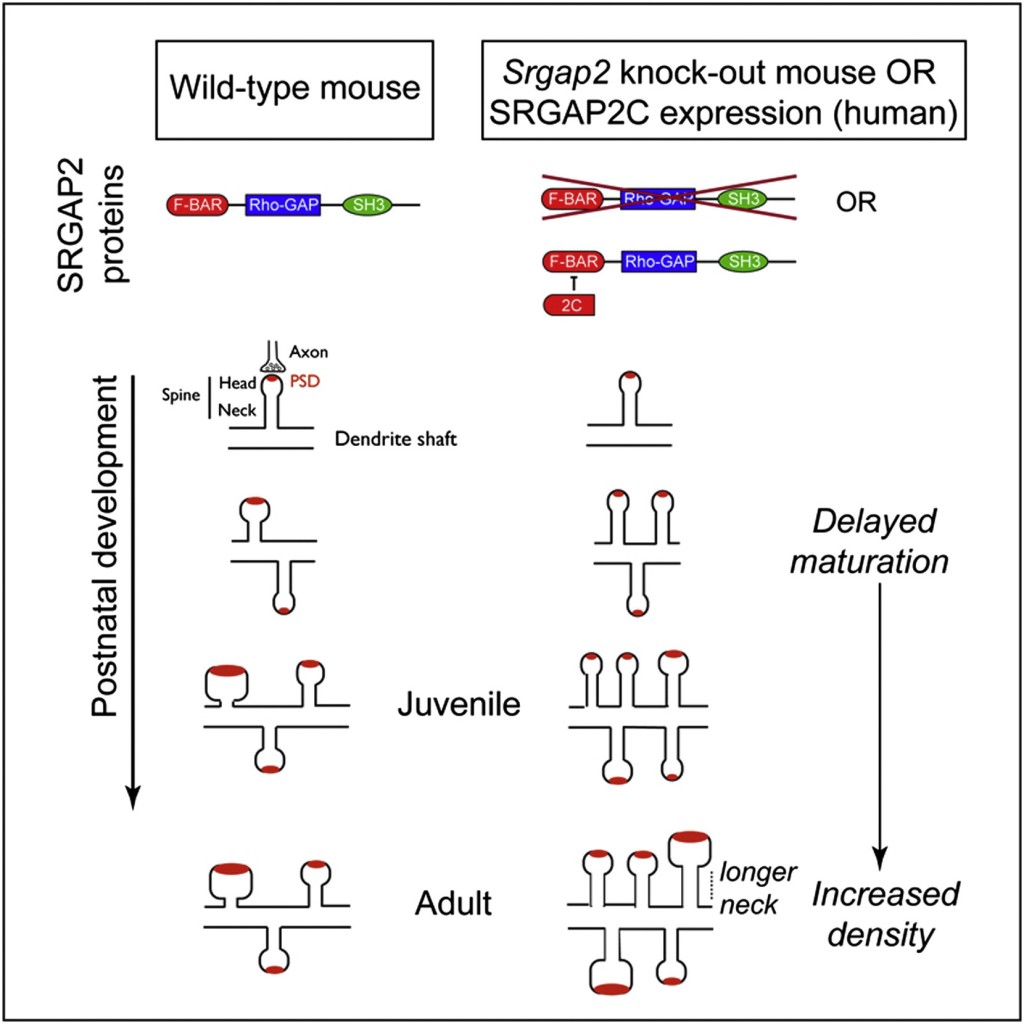
Iniziamo dal titolo del lavoro originale: “Inhibition of SRGAP2 Function by Its Human-Specific Paralogs Induces Neoteny during Spine Maturation”. Cioè: l’inibizione della funzione di SRGAP2 da parte dei suoi paraloghi specifici dell’uomo induce neotenia durante la maturazione delle spine dendritiche. Iniziamo col combattere i paroloni difficili.
La prima cosa che mi sono chiesto è: che diavolo sarà mai la neotenia? Mica sono onnisciente: google sì però [2], ed ecco la risposta in senso lato: “la permanenza di attributi infantili nell’età adulta”. Il resto del titolo è facile: SRGAP2 è un gene umano: si capisce perchè è scritto tutto maiuscolo; lo stesso gene nel topo si chiama ad esempio Srgap2. Considerando un genoma, i paraloghi sono geni che si sono originati da un unico gene ancestrale (per duplicazione), e che sono andati incontro ad evoluzione indipendente.
Il titolo in italiano normale potrebbe suonare: La regolazione di un gene da parte di copie duplicate dello stesso gene 2,5 milioni di anni fa (presenti solo nel genoma umano) attribuisce proprietà uniche ai nervi umani.
In buona sostanza, dai dendriti umani si originano più spine dendritiche per l’azione “rallentante” che le copie sovrannumerarie (e talvolta incomplete) di SRGAP2 hanno sul processo maturativo dei dendriti stessi.
Ora controlliamo un po’ se la faccenda dei paraloghi è vera. Parlando terra terra, sostanzialmente il genoma umano è un file di testo di 3 gigabyte: una parola unica di 4 lettere [3] (spezzata, a ben vedere, da 45 spazi [4]). I server che noi interroghiamo però non conservano solo questa informazione: il genoma infatti è annotato: contiene proprio delle note che dicono al programma informatico che stiamo utilizzando per visualizzarlo (e a noi) dove vengono posizionati gli elementi funzionali. Quando chiedo SRGAP2 al mio genome browser preferito lui sa dove andarlo a pescare proprio perchè il genoma è annotato.
Per verificare che vi siano questi paraloghi, andiamo a pescare in banca dati la sequenza codificante (che informalmente chiamo cds) di questo gene. Faremo così: andremo a pescare la cds di Srgap2 e di SRGAP2 e andremo a cercare sequenze a loro simili nei rispettivi genomi (murino e umano) (Potete sbizzarrirvi andando a cliccare furiosamente voi stessi qui).
Dei numerosi trascritti che si originano dai locus (o come dicono quelli che ignorano la grammatica italiana – a cui sono costretto ad adeguarmi per la moda – dai loci) di Srgap2/SRGAP2, peschiamo semplicemente il primo e diciamo al computer che vogliamo allinearlo contro TUTTO il genoma. Mi ricordo quando facevo questa roba per la tesi: potevo tranquillamente andare a prendere il caffè e a fare merenda; oggi nel giro di mezzo minuto i server avranno controllato qualsiasi appaiamento in qualsiasi orientamento in qualsiasi pezzo del genoma per fornirci la risposta sul piatto d’argento.
Per fare questo utilizziamo BLAST: come dice il suo acronimo, è uno strumento di ricerca terra-terra per allineamenti locali. Selezioniamo il genoma che vogliamo, diciamogli che gli allineamenti che cerchiamo non sono nemmeno fantastici, e lanciamoci in questa ricerca alla cieca.
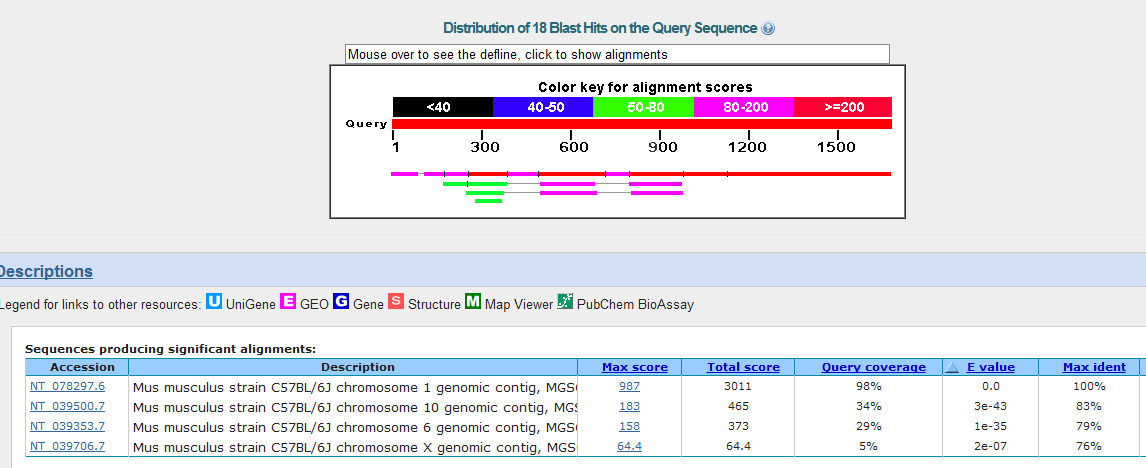
In questa elegante grafica degna dei primi videogiochi, scopriamo che ne è delle sequenze che assomigliano a Srgap2 nel genoma murino.
Vediamo che succede dentro di noi:
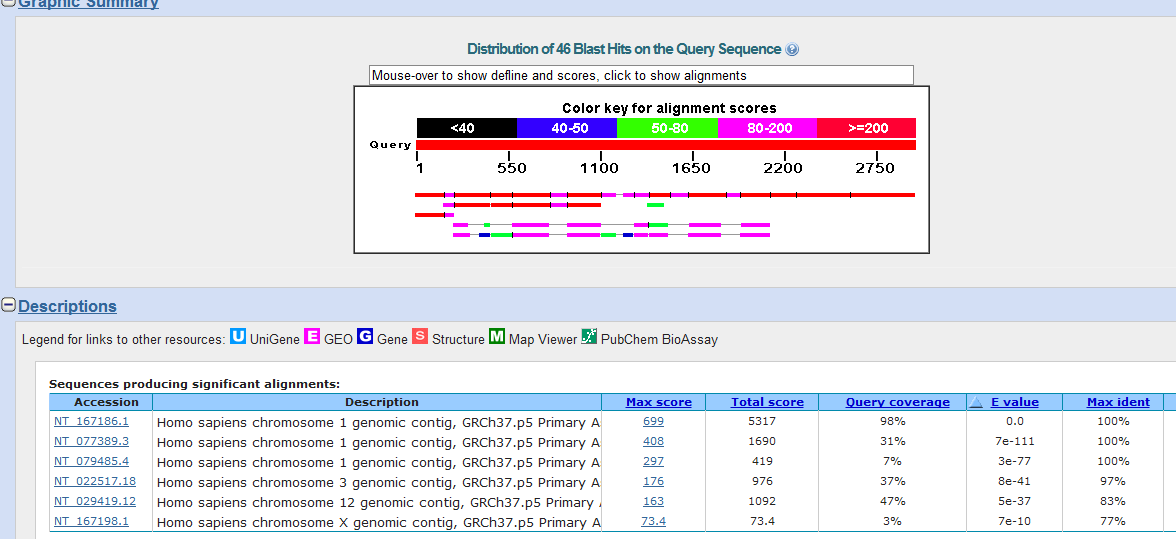
Il numero delle hit nell'uomo è decisamente maggiore: peraltro quelle sul cromosoma 1 sono quelle che cerchiamo. SRGAP2 si trova sul cromosoma 1 sia nell'uomo sia nel topo, e gli eventi di duplicazione ancestrale hanno interessato proprio quella regione.
Utilizzando la pausa caffè e la nostra incrollabile diffidenza, abbiamo verificato “in cieco” (perchè quello che abbiamo cercato non erano dati annotati ma dati grezzi, sequenze nude e crude) quanto asserito dai ricercatori. Certo, nella pausa caffè e alla tastiera non abbiamo fatto anche tutti gli esperimenti che sono serviti per dimostrare tutto il resto… ma l’importante è accontentarsi! :)
Piermatteo
Note a piè pagina
1. Ecco qui la notizia originale
2. Non scherzavo! http://www.thechurchofgoogle.org/
3. A,C,T,G. Tralasciando la 5 metilcitosina (comunque C).
4. Per spezzare il tutto in 46 cromosomi. Ehi, cosa mi sto dimenticando? O:)
Tag:BLAST, cervello, espressione, evoluzione, gene dell'intelligenza, geni, genoma, intelligenza, neuroni, scimmie, scimpanzé, sistema nervoso, uomo
seguici anche su Facebook!